植物共発現データベース「ATTED-II」に解析・考察を支援する新たな解析ツールが実装されました
- その他
- ファンディング
- 統合化推進プログラム
2024年9月27日、東北大学 大学院情報科学研究科の大林 武 教授らは、植物の遺伝子共発現データベース「ATTED-II」に、遺伝子の共発現関係を容易に閲覧・解析するための新たな機能を追加しました。
近年、次世代シークエンサーを用いてさまざまな植物の研究が進められています。ところが、穀物や野菜などの実用植物を含む「非モデル植物」は、シロイヌナズナなどのように実験室内でよく研究されてきた「モデル植物」とは異なり、多くの遺伝子の機能がまだわかっていません。研究者によって様々な研究のアプローチが採られていますが、特定の条件下や環境下で発現量が相関して変動する遺伝子(共発現関係にある遺伝子、共発現遺伝子)のグループを調べることも有用な手段のひとつです。共発現遺伝子は一連の生理的機能をともに担っていることが多く、機能のまとまりや遺伝子の複雑な制御関係の理解に役立つものと期待されるからです。ただ、ひとつの遺伝子が2つ以上の生理的機能を関わっていることも珍しくなく、それを正しく理解するには専門家による複雑な解析が必要でした。
ATTED-IIは2004年11月から開発されています。シロイヌナズナなどのモデル植物を中心に、公開されている主な植物種のトランスクリプトームデータを大規模に解析して収載しており、共発現遺伝子を探索できるデータベースとして広く使われてきました。ところが、遺伝子間の発現プロファイルの類似性を明らかにしても、これまでのATTED-IIに搭載されていた機能だけで、その共発現に関わる条件や環境要因を明らかにすることは、簡単なことではありませんでした。
今回の更新では、共発現関係の主成分(Principal component、以下PC)を分析するツール、「PC View」が新たに実装されました。PC Viewを用いると、着目する2つの遺伝子が共発現する条件や環境を調べることができます。また同じく新たに実装された「CoexViewer」では、着目する2つの遺伝子の発現パターンの相関図を閲覧できます。これら2つのツールによって、共発現遺伝子が担う生理的機能やその制御関係の考察が容易になりました。
上記の新規機能のほか、新たなコムギ、オオムギの情報が追加され、全11植物種の共発現情報を閲覧できるようになりました。また共発現遺伝子の相関関係をネットワークとして表示するNetworkDrawerでは、新たにパラログ情報(KO, KEGG Orthologyが同じ遺伝子)を表示できるようになりました。さらに、遺伝子情報からは、様々な植物種のゲノムやマーカー情報をキュレーションしたポータルサイトのPlant GARDEN (Genome And Resource Database Entry)へのリンクが追加されました。
今回のATTED-IIへの新規機能実装については、2024年10月22日(火)から25日(金)にかけて那覇で開催される1st Asia & Pacific Bioinformatics Joint Conferenceでも発表が予定されています。
ATTED-IIは、JST統合化推進プログラムの研究開発課題「非モデル植物のための遺伝子ネットワーク情報活用基盤(研究代表者 大林 武 東北大学 大学院情報科学研究科 教授)」の一環で開発・提供しています。
<ATTED-IIに収載している植物種(11種)>
- シロイヌナズナ(Arabidopsis)
- アブラナ(Field mustard)
- ダイズ(Soybean)
- ウマゴヤシ(Medicago)
- コメ(Rice)
- ポプラ(Poplar)
- トマト(Tomato)
- ブドウ(Grape)
- トウモロコシ(Maize)
- コムギ(Wheat)(今回新たに収載された植物種)
- オオムギ(Barley)(今回新たに収載された植物種)

PC Viewは個々の共発現関係を理解するためにメタサンプルとして推定できる遺伝子発現データの PC (主成分) を説明するページで、本図は、例としてシロイヌナズナのPC Viewを画面キャプチャしたもの。
PC Viewでは次の4種類の情報を確認できます。(1) サンプルの発現量データに対する主成分 (PC) 分析結果のプロット図。第1から第10までの各主成分に強く関与するサンプルを赤丸・緑丸の表示で確認することができます。(2) 第1から第10までの各主成分に強く関与する、代表的な6つのサンプル (負荷量 (PCA loading) が最も高い3つのサンプル (赤字) と最も低い3つのサンプル (緑字)) が含まれるstudy10件。(3) 発現量が正に相関している上位100遺伝子のリスト。(4) 発現量が負に相関している上位100遺伝子のリスト。リストアップされた遺伝子は、関連した生理的機能や制御関係にある可能性があります。
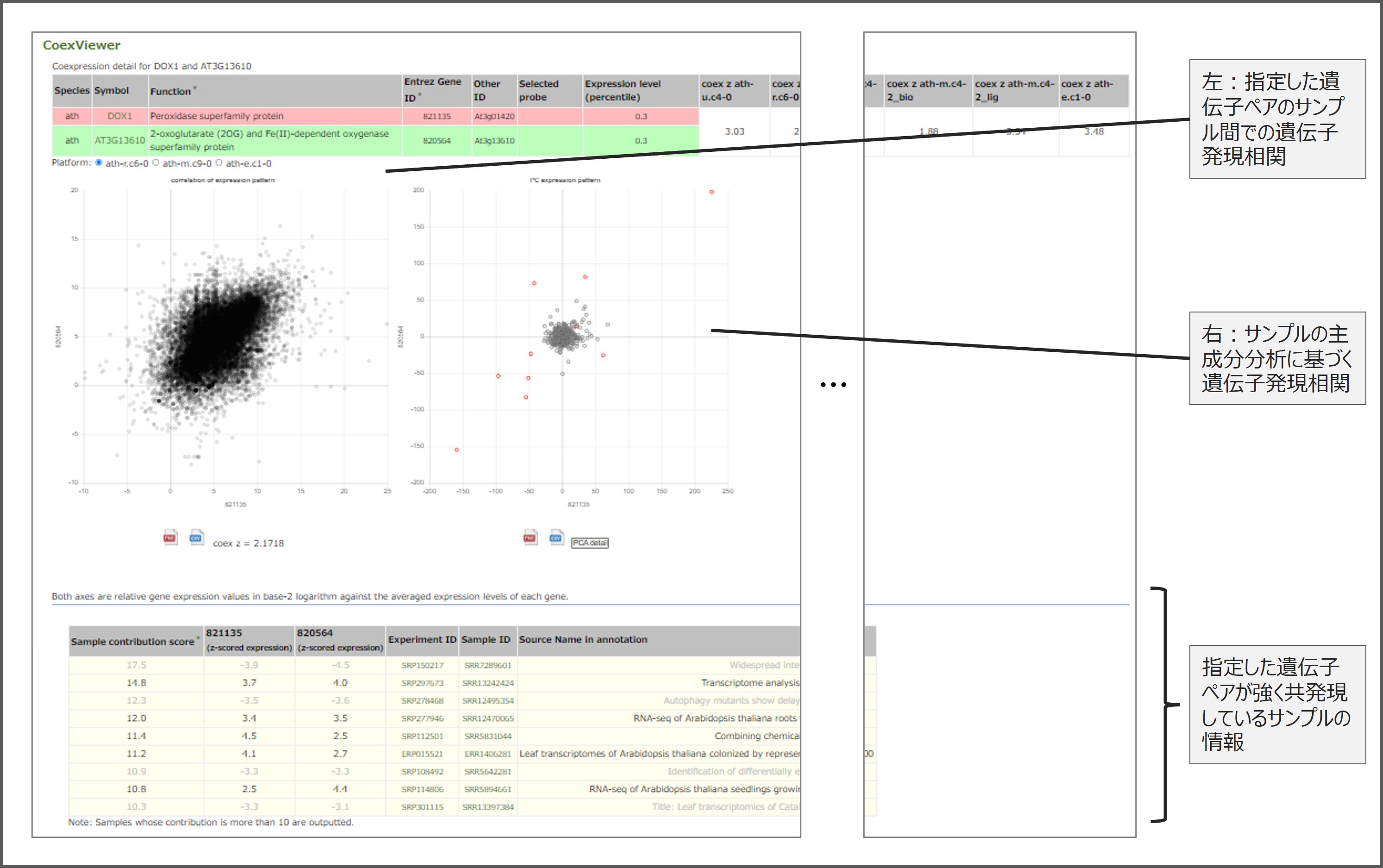
CoexViewerは指定した遺伝子ペアの遺伝子発現相関を表示するツールで、本図は、例として、シロイヌナズナのDOX1(Peroxidase superfamily protein)とAT3G13610(2-oxoglutarate and Fe(II)-dependent oxygenase superfamily protein)についてCoexViewerで表示した結果を画面キャプチャしたもの。サンプル間での遺伝子発現相関に加え、サンプルの主成分分析に基づく遺伝子発現相関も示します。その際、指定した遺伝子ペアが強く共発現しているサンプル主成分を解釈することで、共発現の背景にある生物学的な意味を理解することができます。
関連リンク