生物画像データの標準化で、科学を加速する 国際的な標準ファイル形式「OME-Zarr」の開発 / 理化学研究所・京田 耕司
生物画像データ(イメージングデータ)は生命科学研究に欠かせませんが、その共有・公開は150種を超すファイル形式の存在、1チャネル200GBを超えるほどの大容量化が課題となっています。こうした課題の解決に向け、欧米日が参画する国際コミュニティによって標準ファイル形式「OME-Zarr」の開発が進められています。
今回、2023年7月に出版されたOME-Zarr関連論文の共著者のひとりである理化学研究所の京田 耕司 技師にメール・インタビューを行い、生物画像データ特有の課題、解決に向けた国際的な動向、将来的な期待、京田氏が研究基盤の開発に取り組む動機などを伺いました。

京田 耕司
理化学研究所 生命機能科学研究センター 技師
研究背景――バイオイメージングの利用にまつわる2つの課題と国際的な取り組み
生命科学研究と顕微鏡による観察とは、切っても切り離せない関係にあります。生物の基本単位である細胞の発見は、17世紀、イギリスのロバート・フックによるコルクの顕微鏡観察結果が由来となっています。彼は、コルクが小部屋に分かれていることを見出し、「細胞 (Cell) 」と名付けました。現代においても、細胞・組織の形状やタンパク質の局在の変化を観察するなど、さまざまな生命現象を理解するために、顕微鏡が広く利用されています。
しかし、顕微鏡を使って撮影された画像データの共有・公開には、主に2つの課題がありました。1つ目の課題は、顕微鏡画像データにおける多様なファイルフォーマットへの対応です。これまで、各顕微鏡機器メーカーが独自に開発したものからTIFFなどの汎用的なものまで、150種類を超えるフォーマットが使用されています。これらのフォーマットを読み書きするツール (Bio-Formats *) が広く使用されているものの、その開発・維持にはかなりのコストがかかります。
* Bio-Formats
異なるソフトウェアや研究グループ間で画像データの交換を容易にするために開発された、150種類を超える画像フォーマットの読み書きを可能にするツール。
2つ目の課題は、非常に大きなサイズのデータの共有です。例えば、数十万を超える細胞の挙動をAMATERASという最先端の顕微鏡を使って撮影した画像セットは、1チャネルで200GBを超えています。このような大容量ファイルを研究者が各自の端末にダウンロードすることには限界があるため、データをクラウド上で共有し、可視化・解析することが求められています。しかしながら、生命科学分野での画像データには、クラウドに最適化されたフォーマットがこれまで存在していませんでした。
これらの課題に対処するために、国際的なバイオイメージング・コミュニティでは、共通フォーマットの開発が進められています。統合化推進プログラムでバイオイメージング・データベースSSBDの開発にたずさわる、理化学研究所 大浪チームの京田技師は、国際的なコミュニティの一つである「OME-NGFF(Open Microscopy Environment-Next Generation File Format)コミュニティ」に参加し、クラウドに最適化された次世代のバイオイメージングデータ・フォーマットであるOME-Zarr(オーエムイー・ザール) の開発に貢献しています。2023年7月には、OMEに参加する欧米日の研究者との共著で、OME-Zarr および OME-Zarr に対応するツールやデータリソースの詳細を解説する論文を出版しました。
OME-Zarrが広く採用され、バイオイメージングデータの共有・解析のための環境を容易に提供できるようになれば、世界に散在するデータを統合的に解釈することが可能になり、多くの研究が加速されることが期待されます。
| 名称 | 説明 |
|---|---|
| OME-NGFF | 国際的なバイオイメージングコミュニティにより開発されている画像ファイル形式の仕様のこと。本仕様に準拠したファイル形式の実装として、Zarr形式をベースにOME-Zarrが開発されている。 なお、「NGFF」は、大規模な多次元の生物画像データをクラウド上で効率的に管理・利用するためのファイル形式についての一般的な仕様。 |
| OME-Zarr | 画像ファイル形式の名称。OME-NGFF仕様を満たす実装のひとつ。大規模な多次元の生物画像データをクラウド上で効率的に管理・利用できる。 |
| Zarr | 高次元配列データを格納するためのファイルストレージ形式の名称。一般的に、大規模な高次元のデータのクラウドでの取り扱いに優れる。 |
| TIFF | 画像形式のひとつ。多くのソフトウェアで利用される、汎用的なファイル形式。2次元のデータのローカルでの取り扱いに優れる。 TIFF形式がベースで、メタデータをOME-Zarrと互換性のあるかたちで保管できる「OME-TIFF」も開発されている。 |
誰でも科学に貢献できる世界を願って
Q1: はじめに、先生が研究開発にたずさわることとなった経緯や動機、興味をお持ちのことについて教えてください。
これまで私は、大規模な生命科学データの解析に興味を持ち、研究を進めてきました。大規模遺伝子発現データから始まり、体系的RNAiによる線虫初期胚の細胞核分裂動態データなど、生命科学のさまざまな分野のデータを情報科学の技術を使って解析してきました。このプロセスで、私たちだけで計測したデータを使って全ての知識を引き出すことは難しいと気づき、データを広く共有して利用してもらうことが科学の進展にとって最良だと考えました。
そのためには、フォーマットを統一し、メタデータを付与して、データを利用しやすい形で提供し、国際的な枠組みでデータベースを連携・運用することが重要です。これらのデータの利用が科学者だけでなく一般の人々にも広がり、誰でも科学に貢献できるような世界になることを願って、イメージングデータの共有や解析プラットフォームの開発に日々取り組んでいます。
Q2: バイオイメージングデータの共有・解析における課題は何でしょうか? また、OME-Zarrはそうした課題の解決にどのように寄与するのでしょうか?
特有の課題として、X、Y、Z (空間座標)、T(時間座標)、C(チャネル)といった高次元の情報を持つバイオイメージングデータに対する高速なデータアクセスの実現が挙げられます。この課題についても、OME-Zarrを利用することにより解決することができます。OME-Zarrは、大容量のデータを分割し、制御情報*のついた断片化されたデータ配列に保存することで、高速なデータアクセスを可能にしています。さらに、これらの断片化されたデータ配列を圧縮することもサポートされ、ファイルサイズや転送時間を低減することもできます。OME-Zarrの基盤である Zarrはクラウド利用を前提に開発されたフォーマットなので、データ共有についてはAmazon S3などのオブジェクトストレージを利用できます。これにより、データ共有をサーバーレスで行い、並列データアクセスを可能にすることで、ネットワークを介した高速なデータアクセスが実現できます。
他の課題として、撮影対象が分子から細胞、組織、個体までさまざまであり、多様なイメージング手法が存在することが挙げられます。したがって、その画像データの生物学的意義を理解し、解析に利用するためには、撮影対象やイメージング手法、顕微鏡機器の構成や設定など多岐に渡る情報が必要です。OME-Zarrでは、こうした情報をメタデータとして柔軟に格納できるよう、現在、REMBIやQUAREP-LiMiなどのバイオメタデータのガイドラインに従ったメタデータを保存する仕組みの開発が進められています。
制御情報
画像ファイルの解釈や表示に必須な情報。
Q3: OME-Zarr の実装に向けた開発に関し、苦労された点や印象に残っている点を教えてください。
OME-Zarrの基盤であるZarrは、クラウド時代における新しいフォーマットで、関連情報がまだ少ない部分もありました。しかし、同時に最新の技術に触れることができる興奮や面白さもあります。ストレージに関しても、これまでの経験とは異なるため、調整に苦労しましたが、研究所のシステム部門の協力を得ながら、データの公開と共有が実現しました。実際に100GBのデータを埼玉県和光市のクラウドにアップロードし、兵庫県神戸市からアクセスして、ストレスなくデータを可視化できた瞬間は、少し感動的でした。
OME-Zarrの利点を活かした新たな画像解析技術に期待
Q4: OME-Zarr は、一般の生命科学分野の研究者にとってどのようなインパクトがありますか?
OME-Zarrの利点は、データアクセスのレスポンスが非常に速いことです。従来のTIFFやそれに基づくフォーマットでは、大きなファイルの場合、データを読み込むのに十数分かかることがありました。しかし、OME-Zarrでは、可視化が必要な焦点面や時点のみを即座に読み出すことができます。これにより、napariやFijiなどのツールを利用して画像データの可視化をレスポンス良く行うことが可能になりました。
さらに、OME-Zarrを使用することで、クラウド上でデータの格納・共有、可視化、解析が容易になります。クラウド上での可視化や解析を行うことで、アップロードやダウンロードの手続きを繰り返す必要がなく、複数の場所での利活用や共同研究が実現できます。理化学研究所が運営する統合データベースであるSSBD:databaseでは、OME-Zarrで格納された複数のサンプルデータを公開しています(https://ssbd.riken.jp/ssbd-ome-ngff-samples/)。これらのデータはクラウドストレージ上で共有され、Vizarrなどのツールを使用してウェブブラウザ上で迅速に可視化できます。また、Pythonなどのプログラミング言語を使用して解析することも可能であり、新しい画像解析技術やそれに関連するツールの開発と共有が期待されています。
イメージングデータの共有・解析の環境構築にまつわる研究者の悩みがなくなれば、各々の本質的な研究課題に集中できるようになり、組織・国をまたいだ共同研究の活発化、ひとつのデータから得られる生物学知見の質と量の充実にも寄与するでしょう。
Q5: OME-Zarrの普及によって、どのような未来像が期待できるでしょうか?
上記で説明した OME-Zarr の特徴は、AI技術の開発においても活きてきます。最近のAI技術の進展や深層学習の普及を考慮すると、イメージングデータを使ったさまざまな新たな画像解析手法の開発や加速が期待されます。これに加えて、これまでに開発された画像解析ツールやワークフローを利用することで、画像データから分子や核、細胞を検出し、それらの形態や位置の変化を座標(数値)情報として抽出することが可能になります。
SSBD:databaseの開発・提供を通じ、これまでイメージングデータの共有を促進してきました。今後、画像解析をもサポートするプラットフォームへと拡張させるため、上述のようなツールやワークフローの共有を可能にする予定です。Pythonプログラミング言語で書かれたワークフローや、ELEPHANTなどの汎用性の高いツールを利用可能にし、定量的で客観的な解析が可能になると期待しています。
現状、バイオイメージの解釈には専門家による高度な知見と経験が欠かせませんが、イメージングデータの共有や解析プラットフォームの開発を通じ、例えば、バイオの知識は少ないが画像解析技術に長けた技術者、バイオに高い関心を持つ一般の人々などが参入しやすくなるものと期待しています。誰でも科学に貢献できる、シチズン・サイエンスの実現を願っています。
世界中のデータを繋げることで科学・産業の発展に貢献する
Q6:オープンサイエンスの実現に向け、ファイルフォーマットやデータベースの開発に日本が参画する意義について先生のお考えをお聞かせください。
科学の進展には、競争だけでなく協力も欠かせないと思います。オープンデータやオープンサイエンスの流れを考えると、特に生命科学分野のデータについては、国際的な協力を通じて共有することが重要です。日本の科学や産業を孤立させないためにも、今回のフォーマット開発やデータベースの統合に参加することは、重要な意味を持ちます。データが世界中と繋がりを持たないと、共有しても十分に活用されず、結果的に科学や産業への貢献が低下する可能性があります。日本で計測されたバイオイメージングデータが正当な評価を受けるためにも、ファイルフォーマットおよびデータベースの国際的な連携をこれからも継続していきたいと思います。
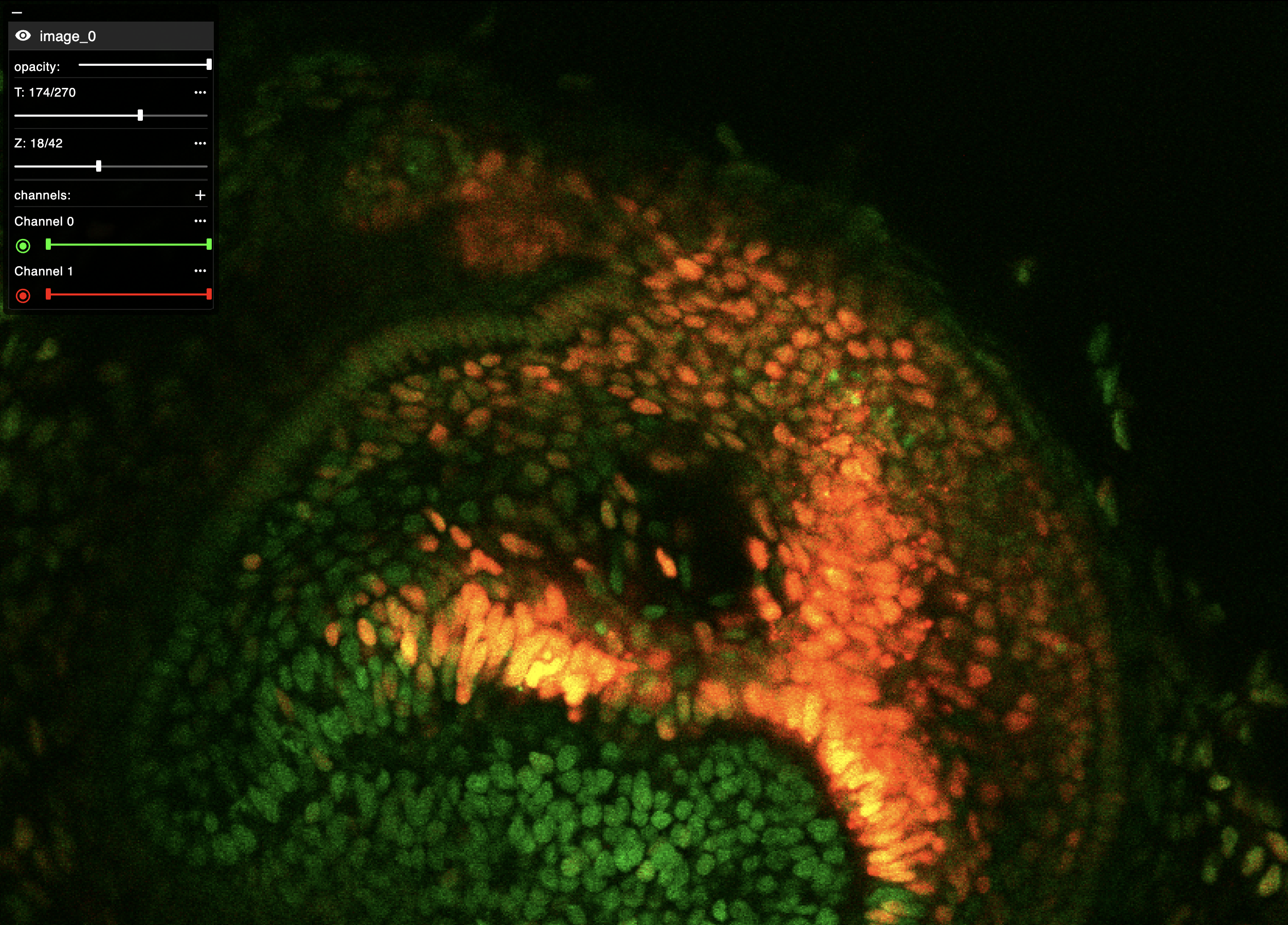
描画ツール「Vizarr」による、OME-Zarr形式画像の表示例。画像の不透明度 (opacity)、時間 (T軸)、深さ (Z軸)、カラーチャネル (本図では緑色、赤色のみ表示) をそれぞれ操作可能。OME-Zarr形式で共有されたデータは、こうしたツールを用いることで、ウェブブラウザ上で簡単に操作・観察できる。なお、表示している画像は、マウス歯形成における上皮細胞の細胞分裂の様子を、ヒストンを緑色の蛍光タンパク質、細胞周期のG1/G0期を赤色の蛍光タンパク質プローブで標識し、共焦点顕微鏡でタイムラプス撮像したもの。