生体組織の立体モデルをぐりぐり動かせる・時間変化を観察できるビューア「QTBD5Viewer」 (開発者インタビュー)
理化学研究所 大浪修一 チームリーダーらは、2023年5月、生命動態データビューア「QTBD5Viewer」を新たに公開しました。このビューアを使うと、 SSBD データベースで公開されている定量的な生命動態の画像を、誰でも自分の PC で自由自在に観察・分析できます。本開発は「統合化推進プログラム」の開発課題の一環として行なわれています。
この記事では、研究に携わった技師の京田 耕司氏、ミランダ ミゲル氏へのメール・インタビュー内容をお届けします。生物学における計測技術や画像処理技術の急速な発展を踏まえたデータ形式、可視化および分析環境の開発、QTBD5Viewer と SSBD データベースの今後の展開などについて 3,000 字に渡って答えていただきました。

京田 耕司
Miranda Miguel (ミランダ ミゲル)
分子や核、細胞の 3D モデルを自由に回転・ズーム
Q1: 今回発表した「QTBD5Viewer」はどのようなソフトウェアでしょうか?
(ミランダ氏) 生命現象の時空間動態データを BD5 形式で記述したファイルを読み込んで可視化するソフトウェアです (BD5 形式については Q3 を参照してください)。
このソフトウェアを利用することにより、 BD5 ファイルに格納された分子や核、細胞といった生物学的な物体の座標情報から 3D モデルを生成し、それらの物体とその動態を3次元空間および時間で観察・解析することができます。3次元空間での観察・解析はマウス操作により行われ、ユーザーは分子や核、細胞といった物体の 3D モデルを自由に回転させ、ズームすることができます。また、スライダーにより時点を変化させて物体の動きをさまざまな角度から観察・解析することができます。インタラクションに対するソフトウェアの応答はリアルタイムに近いため、スライダーで時点を素早く動かすことにより、ショートムービーのように表示することも可能です。
Q2 : どのような方がどのような目的で利用することが想定されますか?
(ミランダ氏) 実験生物学者や計算生物学者など動的な生命現象を解析する研究者の方々が、生命現象を観察して理解するという目的で活用することを想定しています。
また、一般の方々にも生命現象を観察して興味の幅を広げてもらうために開発しました。
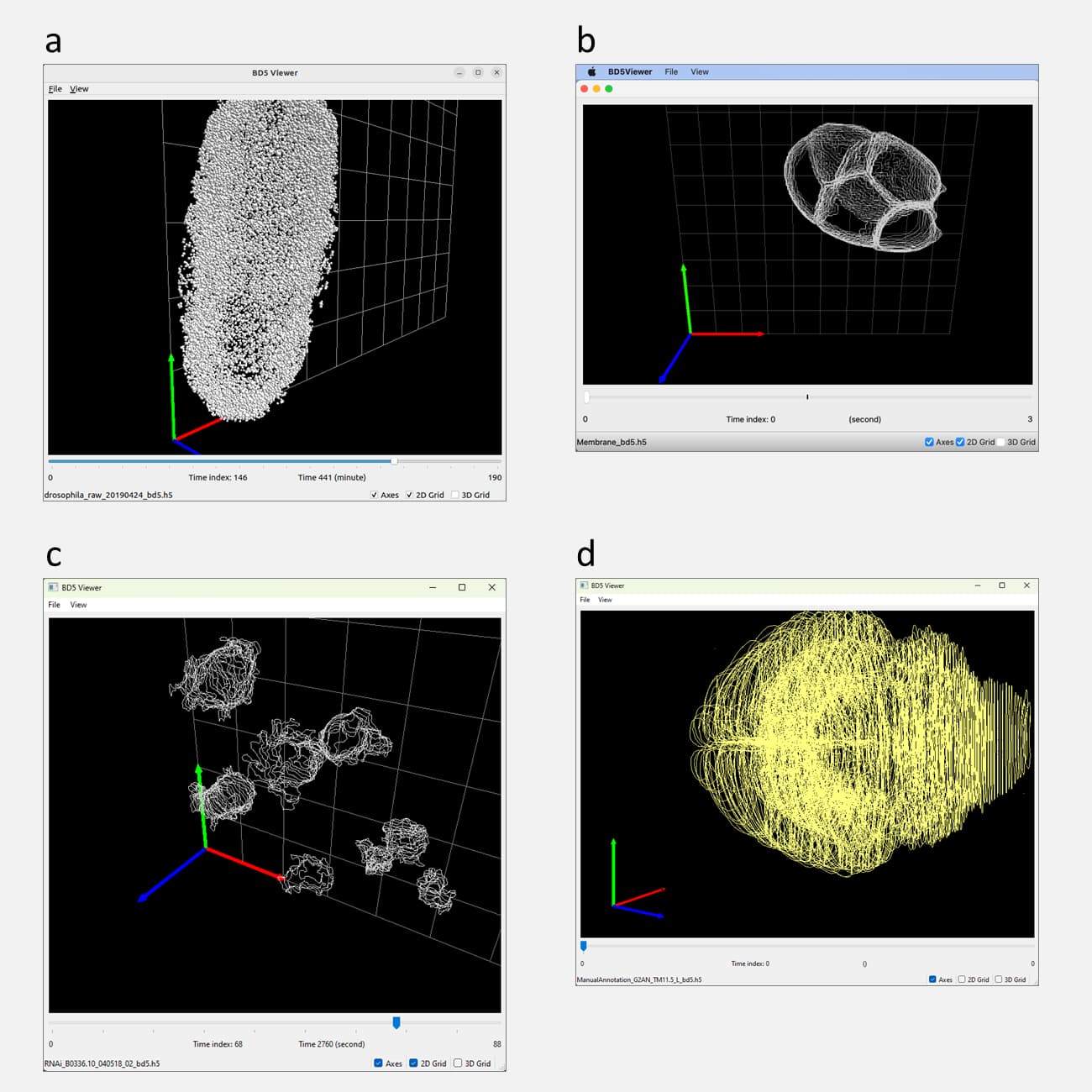
QTBD5Viewerの画面キャプチャ。a) ショウジョウバエ (D. melanogaster) の胚発生における核分裂の可視化、b) 線虫 (C. elegans) 初期胚発生における細胞膜の形状の可視化、c) 線虫 (C. elegans) 初期胚発生における細胞核分裂の可視化、d) マウス (M. musculus) 脳の解剖学的領域の可視化。
生命動態の定量的なデータが増えているが、フォーマットの壁があった
Q3 : 従来の形式の課題と BD5 形式のメリットなども含め、今回、ビューアを開発することになった背景や狙いを教えてください。
(京田氏) ビューアで読み込む BD5 データ形式を開発するにいたった経緯からお話しします。
深層学習を含む画像処理技術が急速に発展しており、ライブセルイメージングの顕微鏡画像から、分子や核、細胞の動態を定量的に計測することが可能になってきました。世界中の研究グループにより、多種多様な生命現象に対して、分子や核、細胞の位置や形状の座標情報とこれらの時間的なつながりが計測されています。しかし、これらを計測したデータは独自フォーマットで記述されているため、解読が困難であったり、個別に入出力や可視化のためのツールを開発する必要がありました。
この問題を解決するため、これらの座標情報と時間的なつながりを統一して記述することが可能なデータ形式である BDML (Biological Dynamics Markup Language) を開発しました。
しかし、BDML はテキストを基盤とした形式であるため、大容量のデータに対する入出力とデータアクセスに問題がありました。
この問題を解決したのが、次に開発した BD5 形式です。BD5 は、アメリカの NPO 法人である HDF グループが開発する HDF5 の採用によりランダムアクセスが可能になったため、必要なデータへのアクセスが劇的に高速化される結果となっています。この BD5 形式で記述されたデータを可視化するための簡単なコードは Jupyter notebook として公開していましたが、インタラクティブに可視化することが可能なツールの開発が望まれていました。分子や核、細胞の動きを理解する際には、時間を行き来して、特に視点を変えながら、それらの動きを観察する必要があるからです。
今回開発した QTBD5Viewer は、これらの操作を行うことができるインタラクティブな可視化ツールとなっています。
Q4 : HDFコンソーシアムと「SSBD」の関係性を教えてください。
(京田氏) HDF コンソーシアムと直接の関わりはありませんが、2022 年の European HDF5 User Group のミーティングに招待され、講演させていただきました。HDF5 のユースケースのひとつとして、コンソーシアムの人たちに興味を持っていただけたのではないかと思っています。
Q5: QTBD5Viewer リリースの反響はいかがですか?
(京田氏) 画像データからの組織形状の定量化を行なっている研究グループから、 QTBD5Viewer を利用したおかげで、データの確認などの作業効率が飛躍的に向上したとご報告をいただいています。このような反響は、我々としても今後の開発のモチベーションにもつながるので、とても嬉しいです。
ブラウザ版ビューアも開発中。より多くの利用を期待
Q6 : QTBD5Viewerの今後の開発計画を教えてください。
(ミランダ氏) 以下の改良と機能追加を行う予定です。
- 動いている各物体の軌跡の表示
- 物体に紐づいたラベルの表示
- マウスやキーボード操作の最適化
- ユーザーエクスペリエンスを向上させるためのカラー利用
このソフトウェアは、C++ 言語と Qt ライブラリで開発されており、ユーザーの OS (macOS, Windows, Linux) に本ソフトウェアをダウンロードして、コンパイルをする必要があります。また、本ソフトウェアをコンパイルする前に、いくつかのソフトウェアの条件を満たす必要があります (手順はマニュアルに詳しく記載されています)。経験の浅いユーザーの中には、インストール作業を面倒に感じる方もいらっしゃるかもしれません。我々が開発・運用している SSBD データベースに格納・共有されているデータに対しては、ブラウザ上で分子や核、細胞といった物体の 3D モデルを生成し、それらの動態を可視化するウェブアプリケーションを提供しています。現在も継続して開発中であり、物体の3Dモデルとそれらの動態の表示、各物体の動きの軌跡や物体に紐づいたラベルの表示のみならず、各種メタデータ (生き物の名前や観察している生物学的プロセスなど) を表示することができるようにする予定です。このアプリケーションは一般的なブラウザ上で動作し、プラグインなどのインストールも不要なため、より多くの方々にご利用いただけるものと考えています。
(京田氏) 現在、バイオイメージングコミュニティでは、関連するデータの格納方法についての議論が活発化しています。特にバイオイメージングデータはサイズが巨大になるため、クラウドの活用に向けた取り組みが行われています。そのひとつとしてバイオイメージングデータの次世代ファイル形式の開発が進められており、 Zarr を基盤とした OME-Zarr という形式が提案されています。我々も、今までの経験を活かして Zarr を基盤とした BD-Zarr の開発に取り組んでいます。今後は、 BD-Zarr に対応したバージョンも開発していきたいと考えています。これにより、ローカルに格納されているデータおよびクラウド上に置かれているデータを可視化することができるようになります。Python を基盤としたバイオイメージングデータの可視化・解析のためのエコシステムである napari を利用することも考えています。
分子や核、細胞の動態の観察を通し、生命の美しさに興味を持ってもらえたら
Q7 : QTBD5Viewer によって、 SSBD の利活用がどのように広がるのか、今後の期待も含めて教えてください。
(ミランダ氏) SSBD データベースは、研究者の方々の協力により、約 700 セット、350GB の BD5 ファイルが共有されています。 QTBD5Viewer の利用により、これまでに蓄積してきたデータの利活用が促進され、その結果、新たな生物学的な知識や洞察が得られることを期待しています。
(京田氏) 実験をされている生物学者の方々に、活用いただきたいと考えています。今まで画像データで観察していた分子や核、細胞といったものの時間変化をより鮮明に捉えることができると思います。また、ソースコードも公開していますので、計算生物学者の方々には、可視化の技術を駆使してさまざまな改良や機能の追加を行なっていただけると考えています。また、教師や学生の方々、さらには一般の方々にも広く利用していただき、分子や核、細胞の動態の観察を通して生命の美しさに興味を持っていただけると嬉しく思います。
Q8 : ほかにも、アピールポイントがあれば教えてください。
SSBD チームは、みなさまの研究活動や教育活動をより豊かなものにするため、引き続き、共有するバイオイメージングデータの充実と、さまざまなニーズに対応したソフトウェアの提供を含むサービスの充実をはかっていきたいと考えています。
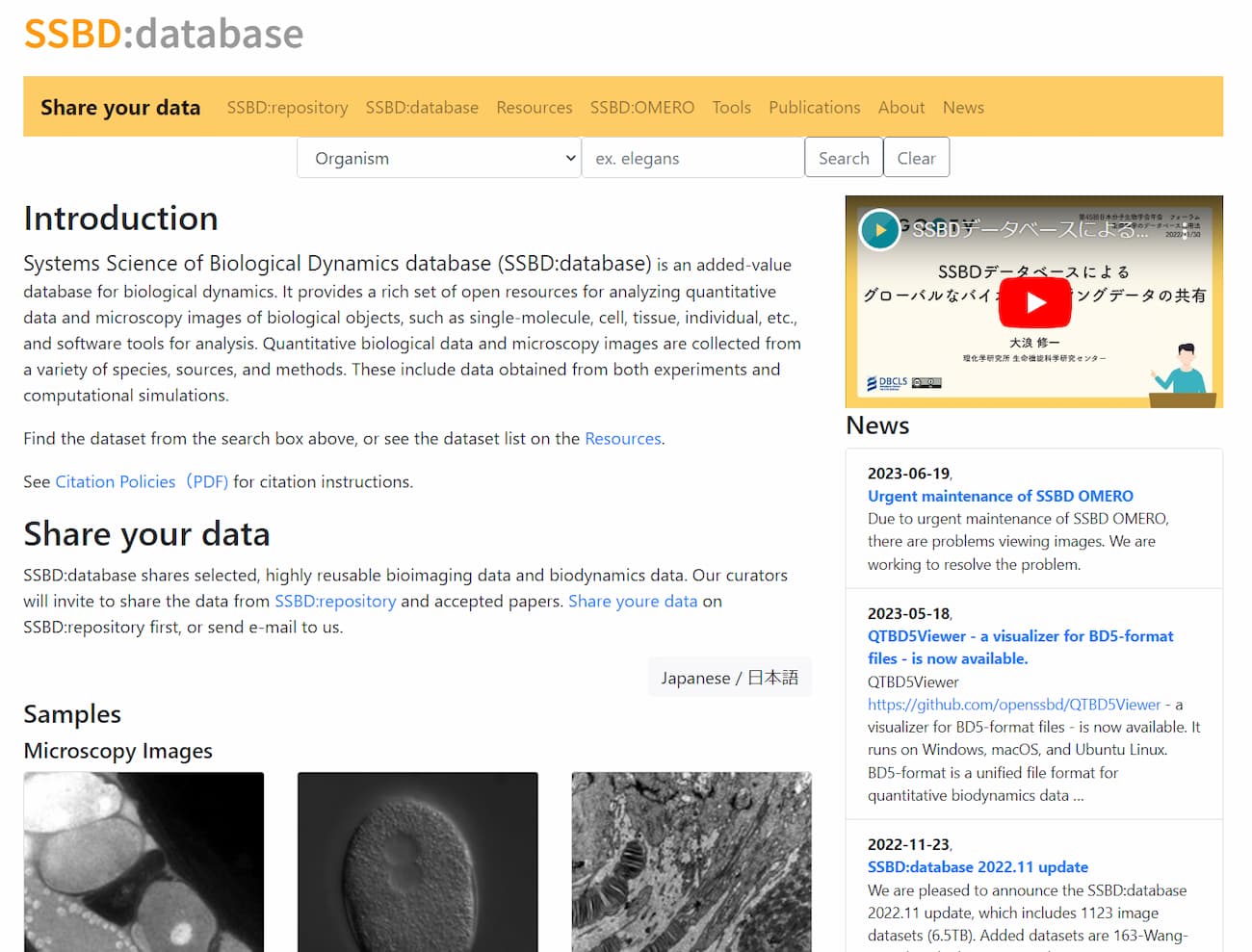
SSBDデータベース。掲載している定量化データは、記事公開時点で約700 件、合計 350GB にのぼる。いずれのデータも ウェブブラウザで閲覧できるほか、今回開発された QTBD5Viewer により、PCにダウンロードして可視化できるようになった。