Bac2Featureを用いて予測された形質情報が、Microbiome Datahubに収載されました
- その他
- ファンディング
- 統合化推進プログラム
京都大学 化学研究所の松井 求 助教らの研究グループは、2025年6月14日、微生物ゲノム配列から微生物の特性や形質を予測するBac2Featureを開発したことを、科学雑誌Bioinformatics Advancesに発表しました。これにあわせて、微生物ゲノムデータベースMicrobiome Datahubに、Bac2Featureで予測した単離菌ゲノムおよびMetagenome-Assembled Genome (MAG)【補足説明】の情報が収載されました。
松井助教らは、標準化された微生物の特性や形質に関するデータセットにさまざまな予測手法を統合し、16S rRNA遺伝子配列から、細胞形態、グラム染色性、胞子形成能や運動性の有無、ゲノム情報、生育条件等の環境特性など、その微生物のさまざまな特徴や性質を予測するBac2Featureを開発しました。論文では、Bac2Featureはその微生物の特性や形質に基づく研究アプローチを可能にし、医学研究などへの応用の可能性が広がると述べています。
一方、国立遺伝学研究所の森 宙史 准教授らが開発しているMicrobiome Datahubでは、2万6千件以上の単離菌ゲノムのほか、さまざまなサンプルのメタゲノムから明らかにされた21万件を超えるMAGを収載しています。Microbiome Datahubでは、Bac2Featureの論文公開にあわせ、Bac2Featureを用いて予測された単離菌ゲノムやMAGが由来する微生物の特性や形質の情報を公開しました。MAGは16S rRNA遺伝子配列を含んでいない場合が多いため、MAGの系統名の情報からBac2Featureで微生物の特性や形質の情報を予測しています。MAGには、実験室では培養できない未知の微生物由来のゲノム配列が多数含まれていることから、Bac2Featureで予測した情報がMicrobiome Datahubで閲覧可能になることで、仮想ゲノムであるMAGが由来する微生物の特性や形質を推定できるようになり、MAGの研究やその利活用が大きく進むことが期待されると森准教授は述べています
Bac2Featureの詳細は、論文「Bac2Feature: an easy-to-use interface to predict prokaryotic traits from 16S rRNA gene sequences」をご覧ください。
Bac2Featureは、統合化推進プログラムの研究開発課題「マイクロバイオーム研究を先導するハブを目指した微生物統合データベースの特化型開発」(研究代表者 情報・システム研究機構 国立遺伝学研究所 森 宙史 准教授)の一環として、研究分担者の松井 求 助教らにより開発されました。
<Microbiome Datahubの収載データ数>(2025年6月5日)
- 単離菌ゲノム 26,076件
- Metagenome-Assembled Genome (MAG) 218,653件
【補足説明】
Metagenome-Assembled Genome (MAG):土壌や環境水、糞便や皮膚組織などから微生物を単離・培養することなく網羅的に解析したゲノム配列を「メタゲノム(Metagenome)」と呼びます。メタゲノムには実験室では培養できない多様な微生物種に由来する断片化したゲノム配列が含まれています。これらの断片配列をコンピュータ上でつなぎ合わせ、元の微生物ゲノムを再現した仮想配列を「Metagenome-Assembled Genome (MAG)」と呼びます。MAGからは、これまで知られていなかった微生物が明らかになるとともに、その微生物が有する新規な酵素遺伝子など、産業応用上においても有用な情報が含まれていることが期待されています。
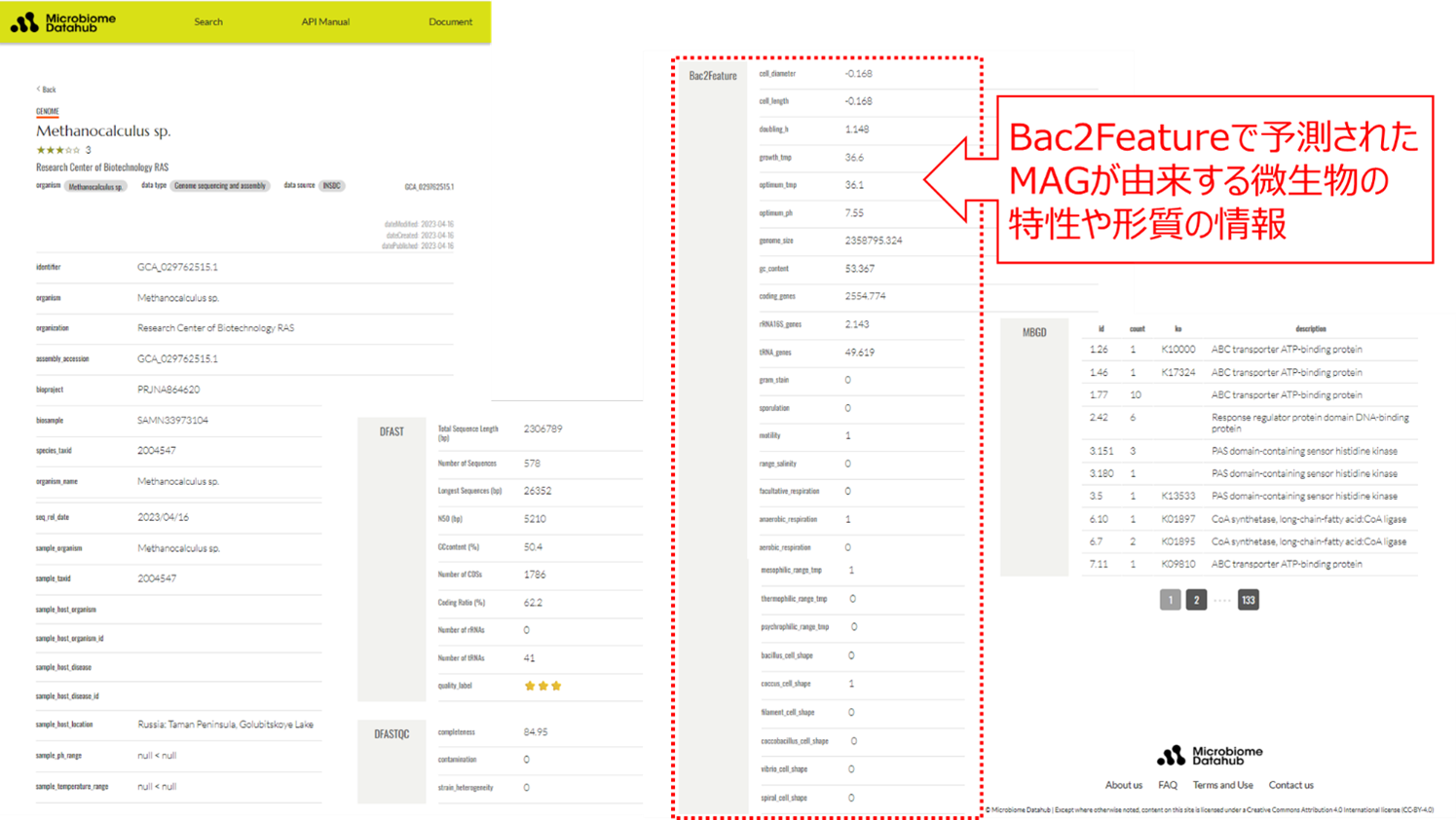
図1 Microbiome Datahubに表示されるBac2Featureで予測された
Metagenome-Assembled Genome (MAG)の特性や形質の情報の一例