空間トランスクリプトミクスデータベースDeepSpaceDB ver.2.0が公開されて高解像度のXeniumデータが新たに収載されました
- その他
- ファンディング
- 統合化推進プログラム
2026年1月16日、京都大学医生物学研究所のVANDENBON Alexis准教授らが開発している空間トランスクリプトミクスデータベース DeepSpaceDB ver.2.0が公開されて、高解像度のXeniumデータが新たに追加収載されました。今回のバージョンアップの詳細については、同日、プレプリントアーカイブbioRxiv に掲載されています。
2024年9月4日に公開されたDeepSpaceDBでは、最も広く使用されているプラットフォームである10X Genomics社のVisiumプラットフォームで調べられたほぼ全ての公開データを収載し、特別なインフォマティクスのスキルを持たない研究者でも、ウェブサイト上で簡単に高度な解析ができるツールを提供し、多くの研究者に利用されてきました。2026年1月16日、VANDENBON Alexis准教授らは、「DeepSpaceDB」のバージョンアップを実施し、Xeniumプラットフォームで取得されたデータを新たに追加収載しました。
Visiumプラットフォームの検出スポットのエリアサイズは直径約55 µmとなっているため、一つのスポットには通常、数個~10個程度の細胞が含まれていると言われています。これに対し、Xeniumプラットフォームでは、細胞内を識別できる解像度で最大5,000遺伝子のRNA分子の位置を直接画像化し、個々の細胞にマッピングするため、一細胞単位の解像度で遺伝子発現を検出できると言われています。一方で、XeniumデータはVisiumデータよりもファイルサイズがはるかに大きくなることから、画像表示やウェブ上でのインタラクティブな解析がとても遅くなるという問題がありました。そこで、VANDENBON Alexis准教授らは、XeniumデータをZarr形式で処理することでアクセスしやすくした上で、Xeniumデータ用の専用インターフェースを新たに開発し、高解像度のXeniumデータの閲覧や解析をユーザーがストレスなくできるようにしました。
DeepSpaceDBでは、Visiumデータと同様、Xeniumデータに対しても、組織切片における遺伝子発現パターンを視覚化する基本的な機能に加え、マウスカーソルで組織切片内の複数の領域を自由に選択してそれらの領域間の遺伝子発現を比較する機能など、インタラクティブなさまざまな解析機能をサポートしています。
Xeniumデータには、DeepSpaceDBのDatabaseタブを開き、サイドメニューのPlatformでXeniumのチェックボックスにチェックを入れるとアクセスできます。
詳しくは、プレプリントアーカイブ "DeepSpaceDB 2.0: an interactive spatial transcriptomics database for large-scale Xenium data exploration "をご覧ください。
<DeepSpaceDBの収載データ数>(2026年1月16日現在)
| 生物種 | Visium V1 | Visium V2 | Xenium |
|---|---|---|---|
| ヒト | 1,020 サンプル | 341 サンプル | 432 サンプル |
| マウス | 648 サンプル | 135 サンプル | 187 サンプル |
| その他 | 0 サンプル | 0 サンプル | 9 サンプル |
DeepSpaceDBは、統合化推進プログラムの研究開発課題「空間オミックスデータ解析用データベースの開発 」(研究代表者 VANDENBON Alexis 京都大学 医生物学研究所 准教授)の一環で開発・提供しています。
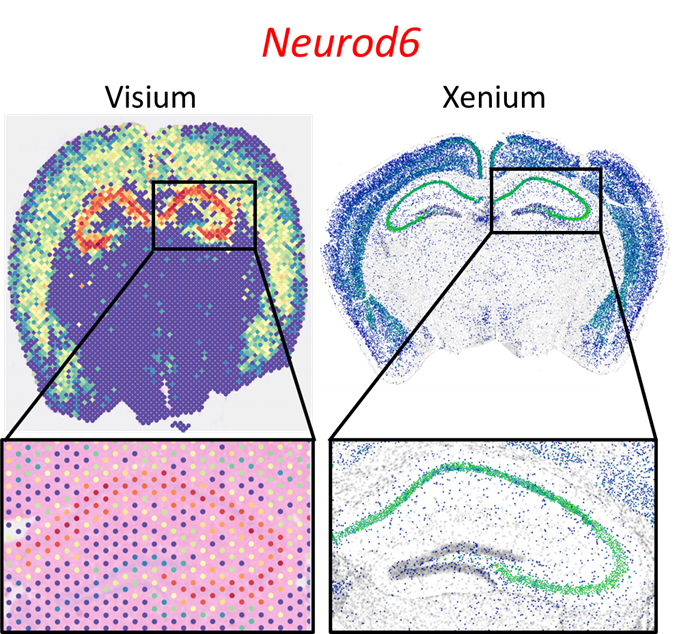
図1: VisiumとXeniumデータの解像度の比較
VisiumとXeniumの解像度の違いを比較した画像。画像は、2つの類似したマウス脳サンプルにおける同一遺伝子(Neurod6)の発現を示す。左の画像はVisiumプラットフォームで、右の画像はXeniumプラットフォーム(右)で取得したもの。
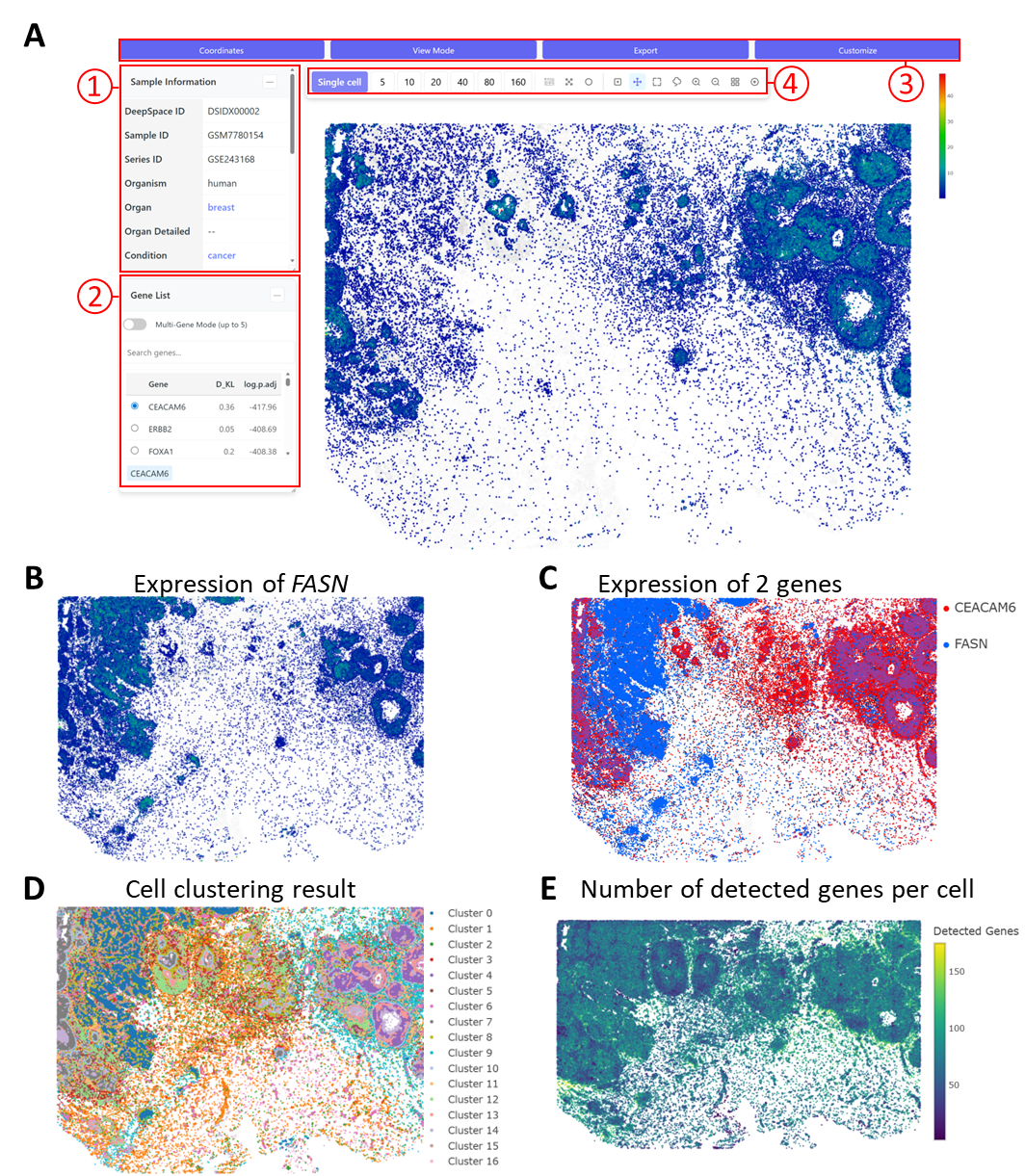
図2: Xeniumサンプルでの任意に選択された領域間の遺伝子発現の比較(DSIDX00002)
A: Xeniumのメインインターフェース ① サンプル情報、② 遺伝子リスト、③ 4つのサブメニュー (「座標」、「表示モード」、「エクスポート」、「カスタマイズ」)、④ ツールバー。遺伝子リストには、singleCellHaystackによって予測された空間パターンの強度順でソートされたすべての遺伝子が含まれる。デフォルトでは、スコアが最も高い遺伝子 (ここではCEACAM6) がプロットされる。
B: もう一つの空間可変遺伝子であるFASNの発現パターン。
C: ユーザーは1つのプロットで複数の遺伝子の発現をプロットできる。このプロットは、CEACAM6とFASNの発現パターンを示す。
D: クラスタリング結果も視覚化可能 「座標」サブメニューで座標を変更することで、サンプル結果をUMAPプロットで表示できる。
E: ユーザーは、細胞あたりの検出された遺伝子数などの品質指標を視覚化することもできる。
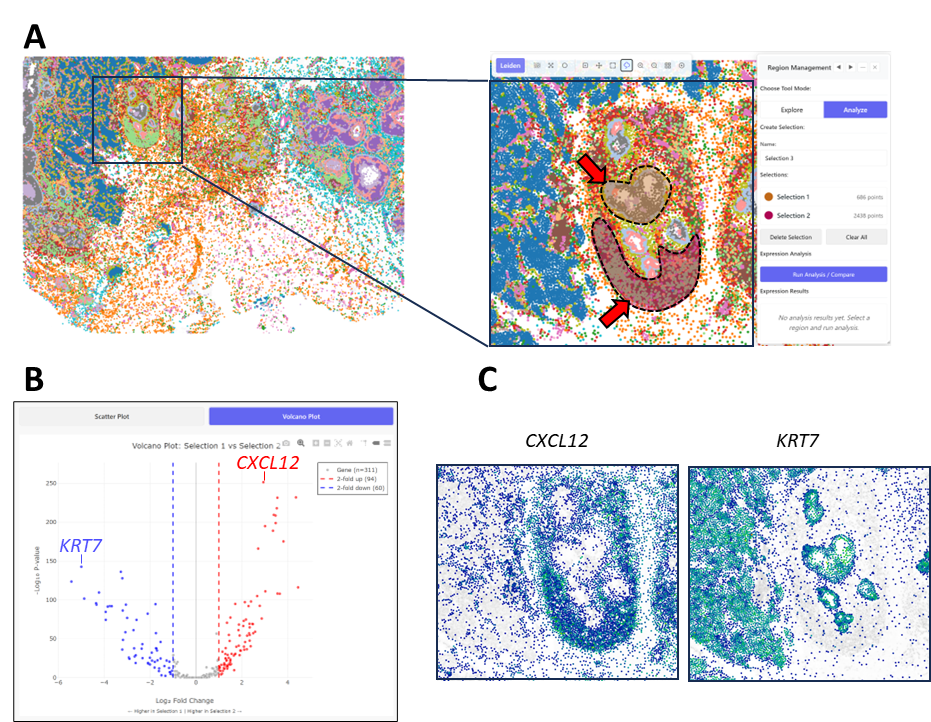
図3: 任意に選択された領域間の遺伝子発現の比較(DSIDX00002)
A: ここではサンプルの一部を拡大表示している。選択ツールを使用して関心領域を2つ選択する(矢印)。「Run Analysis / Compare」をクリックすると、選択した領域の遺伝子発現パターンを比較できる。
B: 選択した2つの領域間の遺伝子発現の違いを示すVolcano plot。この図では、CXCL12とKRT7の2つの発現差のある遺伝子の位置を表示。
C: 2つの遺伝子の発現パターンを可視化し、空間パターンの違いを確認することができる。
用語解説
Visium:10x Genomics社のシーケンスベースの空間トランスクリプトーム解析技術。スライドガラス上の6.5 x 6.5 mmのエリアに密に配置された直径約55 µmのスポットごとに異なるバーコードのオリゴDNAが貼り付けられており、この上に組織片を載せてmRNAを捕捉し、スポットエリアごとの遺伝子発現を計測する。通常、一つのスポットエリアに、数個~10個程度の細胞が含まれていると言われている。
Xenium: 10x Genomics社の高解像度のin situ空間プロファイリング技術。ターゲットmRNAに対して、2つの遺伝子特異的な結合アームと中央の領域を持つ特殊なパドロックプローブをハイブリダイズさせ、それを増幅して蛍光イメージングにより検出するサイクルを複数繰り返すことで、最大5,000遺伝子の発現量を調べることができる。細胞核染色の画像と組み合わせることにより、各細胞における特定の遺伝子の発現量を計測することができる。
Zarr形式:巨大な多次元配列データを効率的に保存・処理するためのデータフォーマット。データを小さな「チャンク」と呼ぶ小さな領域に分割して保存し、必要な部分だけをオンデマンドで読み込むことで、メモリにデータ全体を展開せずとも大規模データを扱えるのが特徴で、大きなデータを扱う生命科学などの分野で利用が広がっている。
関連リンク