第12回「疫学データベースの統合」
はじめに:疫学研究とデータベース
疫学研究は,疾病の罹患をはじめ健康に関する事象の頻度や分布を調査しその要因を明らかにするものであり,古くから疾患の有無を含む健康状態と生活習慣や環境との関係を統計的に明らかにしてきた.近年では,各種のオミクス,すなわち,遺伝子多型や遺伝子発現,代謝産物など分子レベルの因子をくわえた分子疫学研究もさかんに行われており,また,さまざまな研究から得られたデータを統合し,より広範囲で確度の高い情報を得る試みも多く実施されている[1].
疫学研究におけるデータの統合には2つある.ひとつは異なる検体の統合であり,もうひとつは異なるデータ項目の統合である.前者は再現性の確認や検体数を増やしての検出力の向上のために行われ,後者は変数を増やしての正確性や網羅性の向上のために行われる.
異なる検体を統合する方法のひとつは各データセットの集約値の統合,いわゆるメタアナリシスである.これはおもに,PubMed(http://www.ncbi.nlm.nih.gov/pubmed)など文献データベースを活用し,発表された研究論文から解析結果を抽出して実施する.もうひとつの方法は個々のデータの統合である.集約値の統合に比べ柔軟な解析が可能であり,また,詳細な情報を利用できるため精度が高く望ましいかたちの統合である.しかし,とくに終了してから長期間が経過したプロジェクトではデータやプロトコールが逸失していることも多い.
この問題に対し海外では,米国国立生物工学情報センター(National Center for Biotechnology Information:NCBI)のSRA(http://www.ncbi.nlm.nih.gov/sra),GEO(http://www.ncbi.nlm.nih.gov/geo/),dbGaP(http://www.ncbi.nlm.nih.gov/gap)など,データの永続的な管理と研究者間の共有によるデータの再利用を目的としたデータベースの構築がなされた.とくに,dbGaPはプロジェクトを単位として異なるオミクスであっても各検体を関連づけて登録するものであり,疫学データベースそのものといえる.日本国内では,個人の遺伝子多型および表現型の情報レポジトリー(データの貯蔵庫)としてGWAS database(http://gwas.lifesciencedb.jp/cgi-bin/gwasdb/gwas_top.cgi)が構築されており,今後,ほかのオミクスへも拡張される予定である.このほか医療関連では,地域がん登録全国協議会(http://www.jacr.info/)やレセプト(診療報酬明細書)データベース(http://www.mhlw.go.jp/stf/shingi/2r9852000000amvy.html#shingi16)など,公共データベースにおいて法令にもとづき一定の質および量の疫学データが収集されており,いずれも利用申請が受理されればデータを利用することが可能である[2].
しかし,複数のデータベースにまたがった疫学データの統合は容易なことではない.データ項目の標準化が不十分なこと,また,個人の連結がほぼ不可能なことがそのおもな理由である.これを解決するには,標準的な各項目を定義するための情報(メタデータ)の作成と,個人の連結を可能にするような社会制度や情報システムが必要である.
1.疫学データの標準化
疫学データの標準化にあたっては,いずれのオミクスにおいても,どのような項目があるかを洗い出し,用いることのできる標準語彙や仕様があればそれを利用しつつ,各項目を定義するための情報(メタデータ)を策定しその再利用を促すことが必要である.標準にあわずに取得されたデータに関しては,まずは網羅性や精度の高い公共データベースの情報と関連づけておく.こうすることで,何が突き合わせ可能であるかを容易に把握できるとともに,オントロジー(関係データの整理と意味づけのなされたもの)を利用した系統的な集計や解析を実施することができる[3].
ゲノム情報としては,一塩基多型(single nucleotide polymorphism:SNP),コピー数多型(copy number variation:CNV),DNAメチル化などのエピジェネティックな変異情報があり,実験にはSNPチップや次世代シークエンサーなどが用いられている.メタデータを定義する情報は,参照配列(基準となるゲノム配列)とそこでの始点および終点の位置,多型のタイプ(置換,欠失,くり返しなど),変異型の塩基配列などである.参照できる標準的なデータベースとしては,数十塩基以下の短い多型であればdbSNP(http://www.ncbi.nlm.nih.gov/projects/SNP/)があり,rsID(reference SNP ID)が標準IDとなっている.一方,コピー数多型やエピジェネティックな変異情報についてはdbVar(http://www.ncbi.nlm.nih.gov/dbvar)やEpigenomics(http://www.ncbi.nlm.nih.gov/epigenomics)においてデータが参照できるものの,そこでのIDは登録されている実験データに付与されたものであり普遍的ではない点に注意する必要がある.
トランスクリプトームはmRNAなど転写産物の発現情報であり,実験には定量PCR法やSAGE(serial analysis of gene expression)法,遺伝子マイクロアレイ,次世代シークエンサーなどが用いられている.メタデータを定義する情報は測定対象となる転写産物の配列であり,さらに,その参照配列における位置である.ただし,マイクロアレイのような網羅的な実験手法については対象配列の一部だけの測定であったり配列の末端が不明瞭であったりするため,わずかな方法の違いが本質的な差となることもあるので注意が必要である.異なる実験手法あるいは実験キットのデータを統合する場合は,転写産物に付与されたRefSeq(http://www.ncbi.nlm.nih.gov/RefSeq/)などの公共IDを介することが多い.それらは意味的な関係を表わすオントロジーであるGene Ontology(http://www.geneontology.org/)とも関連づけられており,これを介し異なるIDをひもづけることもできる.
プロテオームおよびメタボロームとしては,臨床検査値として得られる代謝産物の情報や,質量分析計による網羅的な解析が代表的なものである.メタデータを定義する情報は対象となる化合物の構造であり,標準的なデータベースにはPDB(http://www.rcsb.org/pdb/home/home.do)やPubChem(http://pubchem.ncbi.nlm.nih.gov/)などがある.実験手法により同定可能な構造の精度には大きな違いがあり,すなわち,化合物群の場合もあれば光学異性体まで判別できる場合もあるため,現在,これらを統合的に関連づけるためのオントロジーの整備が進められている.
表現型および外的因子は,健康状態や疾患への罹患の有無,生活習慣,環境因子などの情報であり,医療をうけた際のカルテの情報や質問票,死亡票,また,被験者からのWebによる直接入力などによりデータを収集する.メタデータを定義する情報はデータの型(順序なしカテゴリー,順序ありカテゴリー,数値など)とその適格条件(カテゴリー型)や上下限値(数値型)であり,どのようなデータであるかの詳細な説明も重要である.病名,薬剤,有害事象などの医療情報であればICD-10(http://www.who.int/classifications/icd/),MEDIS-DC(http://www.medis.or.jp/),MedDRA/J(http://www.pmrj.jp/jmo/php/indexj.php),SNOMED CT(http://www.ihtsdo.org/snomed-ct/)などの標準用語集があり,その名前と定義にもとづくか,可能なかぎりそれらと関連づけておく.質問票により収集される生活習慣および環境因子の情報についてはほとんど標準化がなされておらず,実際,標準化のむずかしい部分である.先行する疫学研究において用いられた質問をなるべく流用し整合性を図ることが望ましい.
2.疫学の統合情報基盤モデル
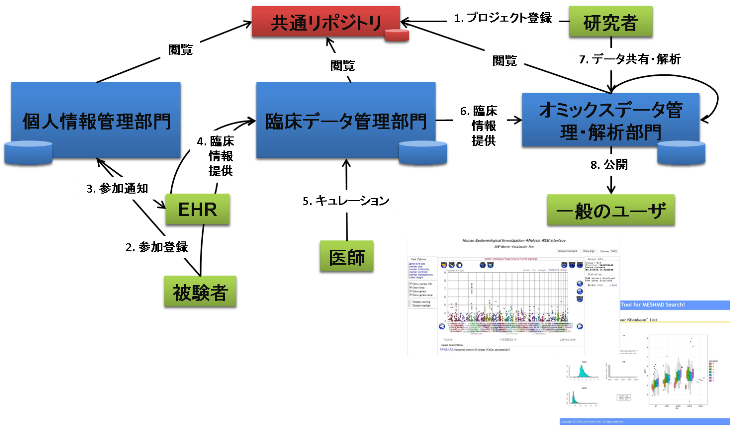
疫学の統合情報基盤をモデル化する際に重要と考えられるポイントとして,1)データ項目の標準化とその利用の推進,2)個人情報と疫学データとの切り離し,匿名化および個人の連結,3)電子カルテからの医療情報の収集,4)収集した医療情報の正誤判断,取捨選択および適切な値への変換(キュレーション)への支援,5)被験者と情報を直接交換するためのインタフェース,6)データアクセス権限の厳格な管理,7)解析手段および集計手段の提供によるデータ活用の迅速化,8)高度なシステムセキュリティ,などがあげられる.現在,筆者らは,ながはま0次予防コホート事業(http://www.city.nagahama.shiga.jp/index.cfm/9,3709,19,158,html)やいくつかの小規模な臨床研究をモデルケースに,生涯健康医療電子記録(electronic health record:EHR)を活用した疫学の情報基盤を構築中である(図1).生涯健康医療電子記録とは複数の病院にまたがる電子カルテや薬局などの処方情報を統合したものであり,疫学研究における利活用が期待されている[4].
この研究では,京都地域連携医療推進協議会の運営するまいこネット(http://www.e-maiko.net/)を利用している.全体は,個人情報,臨床情報,臨床情報およびゲノム情報を管理する3つの組織とそこで共有されるレポジトリーから構成され,この3つの組織はそれぞれ異なる匿名化IDにより検体を扱うことによりデータ漏洩時の被害を極小化している.おのおのの匿名化IDは連結が可能であるが,その対応表は暗号化によりセキュリティを担保する.一般に,電子カルテから得られる情報は文字列であり,また,研究に必要な項目の網羅性も低く,そのままでは解析を行うことはできない.そこで,医師に協力をあおいでデータのキュレーションおよび欠損の補完を実施している.また,Webなどを介し被験者から情報を直接収集する.プロジェクトごとの参加研究者,収集データ項目は共通レポジトリーに登録し,権限の集中管理とデータ項目の標準化を進める.各オミクスデータは承認された研究者間で共有され集中的に解析される.また,研究成果は倫理規定にもとづき被験者個人を特定しない範囲で公開される.このシステムは独立した共通レポジトリーによってデータ項目を管理する部分が肝要であり,これによりメタデータの標準化を進め,異なるプロジェクトを統合的に管理するとともに,将来は個人の健康管理における情報基盤として活用することも可能である.
おわりに
通常の疫学データはプロジェクトごとにデータ項目や検体が一意化および標準化されており,容易には統合できない.ここでは,疫学データを統合する際のオミクスごとの課題や求められる機能を概観し,統合情報基盤を構築する際のひとつのかたちを示した.疫学データは多額の人的および金銭的な資本を投入することで得られるものであり,それらを統合し再利用することにより最大限に活用することは研究者の責任でもある.本稿がより効果的な疫学研究の一助となることを願っている.
参考文献
- 川口喬久, 松田文彦: 個人ゲノム時代のゲノムコホート. 医学のあゆみ, 236, 607-615 (2011) ↑
- 山本景一, 松田文彦: 医療情報の統合に向けた臨床情報データベースの構築. 実験医学, 29, 2501-2507 (2011) ↑
- Pesquita, C., Faria, D., Falcao, A. O. et al.: Semantic similarity in biomedical ontologies. PLoS Comput. Biol., 5, e1000443 (2009) ↑
- Kohane, I. S.: Using electronic health records to drive discovery in disease genomics. Nat. Rev. Genet., 12, 417-428 (2011) ↑
↑ 押下で本文に戻ります。

Licensed under a Creative Commons 表示2.1日本 license ©2012 川口喬久(京都大学大学院医学研究科附属ゲノム医学センター)、松田文彦(京都大学大学院医学研究科附属ゲノム医学センター)
なお、本記事は細胞工学2012年4月号掲載の原稿を改変したものです。