第9回「ヒトゲノムバリエーションデータベースの開発」
はじめに
ヒトゲノムのバリエーション(variation)には,一塩基もしくは数塩基レベルの置換,挿入,欠失と,構造多型もしくは構造異常とよばれる数百塩基から数万塩基レベルの逆位,転座,欠失,重複などがある.本来,多型(polymorphism)とは,集団にマイナーなアリルが1%以上の頻度で存在する状態をいう.
高速大容量のSNP(single nucleotide polymorphism,一塩基多型)タイピング技術とDNAシークエンシング技術の長足の進歩により,ヒトゲノムのバリエーションの解析能力はここ5年ほどで飛躍的に向上した.DNAチップを用いたSNPタイピング技術では,現在,ゲノムの全域に分布する数十万種から数百万種のSNPを解析することが可能となっている.これにより,common disease common variant説(ありふれた疾患には頻度の高いゲノム変異が関与するという説)のもと,数千から数万の検体を対象としてゲノムの全域より疾患に関連するSNPを探索する研究(genome-wide association study:GWAS,全ゲノム関連解析)が世界的に実施され,多くの疾患関連SNPが発見されている.一方,このような解析法では既知のSNPしか対象とならないため,集団において疾患感受性変異の頻度が低いと予想される希少疾患の場合には疾患家系のゲノムをシークエンシングする方法が有効である.現在,全ゲノム領域もしくは全エキソン領域のシークエンシングにより疾患原因遺伝子や疾患原因変異の発見がつぎつぎと報告されている.また,HUGO(Human Genome Organisation,ヒトゲノム解析機構)のPan-Asian SNP Consortiumによるアジア系諸集団の大規模なSNPの解析,1000人ゲノムプロジェクトなどの大型プロジェクトの成果として,集団のあいだのゲノムの多様性も明らかになりつつある.ヒトゲノムの解読完了から約10年をへて,ようやくゲノムと表現型との関係,集団のあいだのゲノムの多様性や変異にはたらいた自然淘汰の過程が明らかになりつつあるといっても過言ではない.近年,これらのバリエーションの情報が急激に増加しているため,系統だった情報の整理と研究者のあいだでの共有は喫緊の課題となり,それらに対応したかたちで多様なデータベースの整備が進んでいる.
ヒトゲノムのバリエーションに関係する代表的なデータベースを示す(表1).集団を考慮したかたちで一塩基もしくは数塩基レベルでの変異を網羅的に収載したデータベースとしては,dbSNPが事実上の世界標準となっている.このdbSNPはマイナーアリルの頻度にかかわらずデータを収載しているため,マイナーアリルの頻度が1%以下の変異も含まれている.また,構造多型に関しては,DGVaが米国NCBI(National Center for Biotechnology Information)のdbVar,および,欧州EBI(European Bioinformatics Institute)のEGAと連携して,アクセッション番号の発行とデータの保管を行っている.DGVaの健常者データを収載したデータベースがDGVである.
一方,変異と疾患との関係を収載しているデータベースとしてもっとも有名なのがOMIMであり,遺伝子疾患に関して臨床的な特徴とその関連遺伝子の変異についておもな知見を参考文献とともにまとめている.また,HGMDは臨床的な特徴などの情報はないが,塩基レベルおよびアミノ酸レベルでの変異の情報までを文献から収載している.収載されている疾患関連の変異情報は多いが,最新版はアカデミアに対しても有料である.また,DECIPHERは,染色体不均衡と疾患との関係を収載しているデータベースである.LSDBはHuman Genomic Variation Societyが維持運営しているデータベースで,事実上,特定の遺伝子もしくは疾患に特化した変異と疾患との関係を収載したデータベースのリンク集となっている.特定の疾患に特化したデータベースには実験を行った集団ごとの情報もまとめられていることが多いが,広範な疾患や変異を取り扱うデータベースには集団の情報がまとめられていない場合が多い.多くの疾患について集団のあいだの共通性と異質性が知られており,この点については整備が不十分である.
表現型と変異との関連についての情報に関して,ここ5年ほどで大規模かつゲノムワイドな疾患関連遺伝子の探索プロジェクトが多数進行したことにより,それ以前には存在しなかった膨大なゲノムバリエーションデータが産生されている.これらのデータ量の膨大さと情報の貴重性から,データの再利用のための研究者のあいだでのデータの共有や,個別の疾患に関連する研究の発展といった観点から,解析結果の公開についての重要性が認識され,これに応えるかたちで,米国NCBIのdbGaP[1]と欧州EBIのEGA(The European Genotype Archive: Background and Implementation, white paper, 2007)が構築された.これらのデータベースには,数百から数万の検体を対象とした症例対照関連研究やコホート研究などの成果だけでなく,比較的少ない検体数で小規模に行った解析,また,次世代シークエンサーによる疾患関連変異の探索結果などが個々の研究ごとにまとめられている.dbGaPには米国NIHの研究費をうけたプロジェクトのデータが,EGAには欧州Welcome Trustの研究費で実施されたプロジェクトのデータが,おもに蓄積されている.EGAでは統計解析の結果を利用するためにも申請が必要であるが,dbGaPではデータの公開は各スタディの公開指針により若干異なるものの,症例対照研究におけるHardy-Weinberg平衡検定値,各モデルでのp値,オッズ比の信頼区間などの計算結果が公開されている.以前は,遺伝子型およびアリル型の頻度データも公開されていたが,頻度の情報であっても技術の進歩により研究参加者の特定にいたる危険性が完全には否定できないとの理由で[2],現在では利用申請が必要となっている.実験の生データや個体レベルの遺伝子型のデータはdbGaP,EGAともに制限付きアクセスの対象となっており,研究目的や倫理委員会における当該研究の承認についての書類などをそえて利用申請をしたのち,データアクセス委員会において審議され許可された場合のみ利用が可能である.また,生データの預け入れや再配布のサービスはないが,全ゲノム関連解析の結果を広く収集しているデータベースにHGVbaseG2Pがある.
わが国の統合データベースプロジェクトにおける取り組み
わが国においても,SNPの解析による全ゲノム関連解析がここ5年間ほどで多く実施されたことをうけ,データの半永続的な管理およびデータの再利用のための研究者のあいだでのデータの共有のため,2007年度~2010年度に実施された文部科学省 統合データベースプロジェクトにおいて,筆者らは,GWAS-DBおよびCNV-DBを構築した.また,データアクセス委員会も発足させ,データの共有化を行うための枠組みを構築し,現在もひきつづき運営している(https://gwas.biosciencedbc.jp/index.Japanese.html).
このデータベースにおける全ゲノム関連解析のデータ公開の基準は,1)遺伝統計計算の結果と遺伝子型およびアリル型の頻度データは公開.ただし,頻度データを一括ダウンロードする際には倫理審査をへない簡易申請が必要.2)個体レベルでのCNV(copy number variation,コピー数多型)の情報は簡易申請ののち利用が可能.3)遺伝子型データおよび実験の生データについては,研究目的などを明記したデータアクセス申請書を提出し,データ共有審査委員会にてデータ使用の可否が審議される.許可が下りた申請者は,第三者への提供の禁止,個人の特定につながる行為の禁止などの条件のもとでデータの使用が可能となる.一方,データを提供する際には,データ共有審査委員会にデータ提供申請書を提出し,検体の提供者から同意書を得ているかなどの審議をへる必要がある.
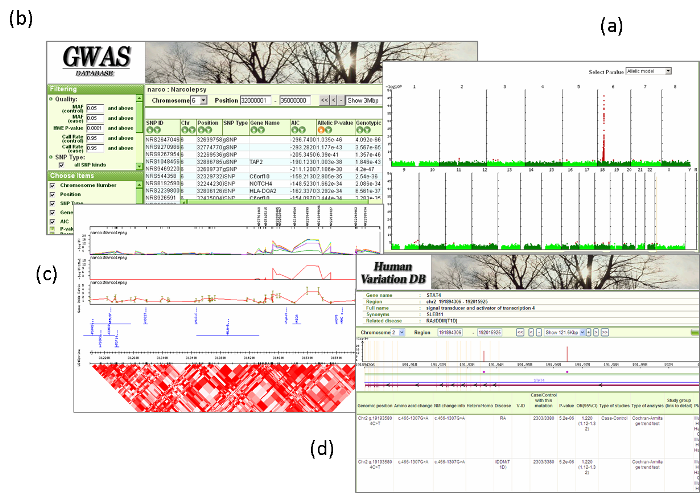
GWAS-DBおよびCNV-DBとも,データ提供者が解析したデータの登録も可能であるが,データベースの側では,SNPごとの品質,検体ごとの品質,集団としての品質,の3項目について品質基準を通過したデータを用いて統計解析を実施している.GWAS-DBには,研究デザインとともに,遺伝子型およびアリル型の頻度の情報,Hardy-Weinberg平衡検定値,傾向検定や劣性モデル検定など各種のモデルでのp値,多重検定の補正結果,ハプロタイプ(単一の染色体におけるアリルの組合せ)でのp値,連鎖不平衡のLODスコア,SNP間の相互作用解析などを登録している(図1).また,SNPの種類,ゲノムにおける位置情報,非コードRNAの情報,CNVの領域などの情報も登録している[3].量的形質の関連解析の結果の登録も可能である.また,全ゲノム関連解析を実施したDNAマイクロアレイのプローブの強度情報を用いて同時にCNV解析を行うことも可能であるが,SNP解析よりもCNV解析のほうが偽陰性や偽陽性を生じやすいため,その結果には注意が必要である[4].
図1.それぞれのデータベースの画面の例
(a)GWAS-DBのマンハッタンプロット(SNPのp値を縦軸に,染色体における位置を横軸にプロットしたもの).
(b)統計解析の結果.
(c)領域図.
(d)ヒトゲノムバリエーションデータベースの領域図.
ヒトゲノムのバリエーションデータベースへの拡張にむけて
次世代シークエンサーにおける技術革新により,時間的およびコスト的にも全ゲノムあるいは全エキソンのシークエンシングによる新規の疾患関連変異の探索が可能となりつつある.現在は,コストやスループットの観点からシークエンシングできる検体の数がかぎられること,また,実験精度の問題も残ることから,難治性疾患あるいは希少疾患における単一遺伝子のエキソン領域での変異の探索が主流である.しかし,低価格大容量化と精度の向上により徐々にそれ以外の疾患も適用されつつある.1検体あたりゲノムの0.1%にあたる約300万のSNV(single nucleotide variation,一塩基変異)が検出されること,また,その約1割はまだdbSNPに未収載であることから,これらのデータの散逸を防ぐためにもこれらをデータベース化し,研究者のあいだで情報を共有するしくみを整備することが重要である.したがって,2011年度に開始されたバイオサイエンスデータベースセンターによる統合化推進プログラムにおいて,全ゲノム関連解析だけでなく次世代シークエンサーおよびそのほかの実験手法により発見された変異と疾患との関連性について収載し,疾患と変異との関係,および,疾患のあいだの関係を概観できるよう,GWAS-DBをヒトゲノムバリエーションデータベースに拡張している(図1).いずれのデータベースも公開用と内部用の2つを備えており,1年間は内部用として運用することが可能であるため,多施設共同研究の公開前の情報共有の場としても利用が可能である.全ゲノム関連解析,全ゲノム解析,エキソーム解析を実施している研究グループにはぜひともその成果の登録をお願いしたく,gwas[AT]lifesciencedb.jp ([AT]を @ にかえてください) にお問い合わせいただきたい.
デンマークのFaroe島の全住民5万人のゲノムをすべて解読し健康記録とリンクさせ将来的に診断に役立てる計画,同様の英国における50万人のゲノムコホート計画,中国Kadoorie Biobankの50万人のゲノムコホート計画など,大規模なゲノム解析と健康記録および電子カルテとのリンク付けによるゲノム,生活習慣,疾患のあいだの解析が実施されつつある.今後,このような情報は飛躍的に増大すると予想され,個別化医療の実現を加速すると思われる.日本人がヨーロッパ系集団やアフリカ系集団などとは異なる薬物応答性変異,ウイルス耐性変異,疾患感受性変異をもつことから,日本人に特徴的な多型や変異と疾患などの関連情報を整理および体系化していくことが個別化医療の実現のために必須である.
参考文献
- Mailman, M. D., Feolo, M., Jin, Y. et al.: The NCBI dbGaP database of genotypes and phenotypes. Nat. Genet., 39, 1181-1186 (2007) ↑
- Homer, N., Szelinger, S., Redman, M. et al.: Resolving individuals contributing trace amounts of DNA to highly complex mixtures using high-density SNP genotyping microarrays. PLoS Genet., 4, e1000167 (2008) ↑
- Koike, A., Nishida, N., Inoue, I. et al.: Genome-wide association database developed in the Japanese Integrated Database Project. J. Hum. Genet., 54, 543-546 (2009) ↑
- Koike, A., Nishida, N., Yamashita, D. et al.: Comparative analysis of copy number variation detection methods and database construction. BMC Genet., 12, 29 (2011) ↑
↑ 押下で本文に戻ります。
表説明
| 名称 | URL | 概説 |
|---|---|---|
| dbSNP | http://www.ncbi.nlm.nih.gov/projects/SNP/ | SNPのデータベース.網羅性はもっとも高いが品質の管理は少しゆるい.ヒト以外の生物種も充実 |
| mutaDB | http://www.mutadatabase.org/ | 突然変異のレポジトリー型のデータベース |
| DGV | http://projects.tcag.ca/variation/ | ヒト健常者のデータのみを収載した構造多型のデータベース.DGVaが多型のアクセッション番号を発行し,dbVarおよびEGAとのデータの交換を行っている |
| dbVar | http://www.ncbi.nlm.nih.gov/dbvar/ | 構造多型のデータベース.疾患情報,臨床情報およびヒト以外の情報も含む |
| DECIPHER | http://decipher.sanger.ac.uk/ | 染色体不均衡と疾患との関係をまとめたデータベース |
| dbGap | http://www.ncbi.nlm.nih.gov/dbgap/ | 実験の規模と手法にかかわらずバリエーションと表現型との関係に関する情報を登録.米国NIHの研究費をうけたプロジェクトを中心に,実験データの預け入れと再配布(審査あり)も行っている.進行中のプロジェクトの情報もある |
| EGA | https://www.ebi.ac.uk/ega/ | 実験の規模と手法にかかわらずバリエーションと表現型との関係に関する情報を登録.実験データの預け入れと再配布(審査あり)も行っている.欧州Welcome Trustの研究費で実施されたプロジェクトのデータを収載 |
| Gen2phen | http://www.gen2phen.org/ | 実験の規模と手法にかかわらずバリエーションと表現型との関係に関する情報を登録 |
| Human Genome Variation DB | http://gwas.biosciencedbc.jp/ | 筆者らが開発しているデータベース.実験の規模と手法にかかわらずバリエーションと表現型との関係に関する情報を登録.実験データの預け入れと再配布(審査あり)も行っている |
| OMIM | http://www.ncbi.nlm.nih.gov/omim | メンデル型の遺伝疾患とその原因遺伝子および変異情報をまとめたデータベース |
| HGMD | http://www.hgmd.org/ | 変異と疾患との関係をまとめたデータベース.商用ではあるが,アカデミアに対し古いデータは無料で公開 |
| KMDB | http://mutview.dmb.med.keio.ac.jp/MutationView/jsp/index.jsp | 変異と疾患情報との関係を収載したデータベース |
| LSDB | http://www.hgvs.org/dblist/glsdb.html | Human Genomic Variation Societyが特定の疾患もしくは特定の遺伝子に特化したデータベースの情報をまとめたサイト |
| COSMIC | http://www.sanger.ac.uk/genetics/CGP/cosmic/ | Sanger Instituteで実施されたがんの体細胞変異のデータを収載したデータベース |
| ICGC | http://dcc.icgc.org/ | 国際がんコンソーシアムで発見された体細胞変異のデータベース |
| 1000genome | http://www.1000genomes.org | 1000人ゲノムプロジェクトによる変異を収載したデータベース |

Licensed under a Creative Commons 表示2.1日本 license ©2012 小池麻子(日立製作所 公共システム事業部)、徳永勝士(東京大学大学院医学系研究科 人類遺伝学分野)
なお、本記事は細胞工学2012年3月号掲載の原稿を改変したものです。