第6回「メタボロームデータベースの開発:メタボロミクスからのゲノミクスの展開」
はじめに
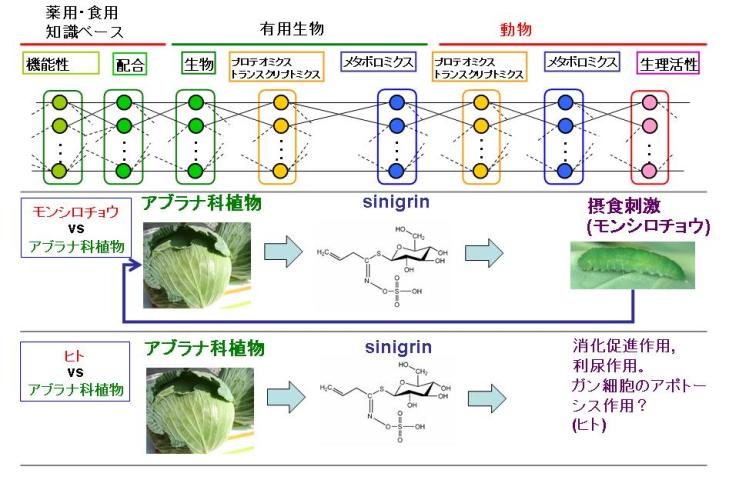
ゲノミクスに代表されるオミックス(トランスクリプトミクス,プロテオミクス,メタボロミクス)と,これまで人類が培ってきた薬用植物および食用植物の知識をリンクさせると,図1のように表現できる.この図にしたがって,代謝産物の総体であるメタボロームを体系化しデータベースを構築すること,さらには,ほかのオミックスと関連づけることにより人類に貢献することが,筆者らの"メタボロームデータベースの開発"プロジェクトの課題である.
図1.オミックス生物学と伝統知識の融合
薬用あるいは食用における伝統知識により,漢方などの生薬の配合法は専門知識として整理されている.配合される生薬のそれぞれはゲノム情報にもとづき種々の代謝産物を生合成する.一方,ヒトは薬用生物あるいは食用生物を摂食し恒常性を維持する.すなわち,薬用生物あるいは食用生物のもつ種々の代謝産物を摂取し吸収することになる.そこで,薬用あるいは食用に関する知識ベースから,有用生物のオミックスとヒト(動物)のオミックスとは複雑に関係し,ヒトの恒常性が維持されている.メタボロミクスから種々のオミックス研究への展開は,いかに有効な代謝産物を有用生物に生合成させるかという問題において,有用生物のゲノミクスと関連する.一方で,摂食した代謝産物をもとにヒトの恒常性は維持されていることから,ヒトのオミックスは恒常性の理解という観点からも研究されており,そのなかでヒトのメタボロミクスに焦点をあてることも重要となっている.
たとえば,モンシロチョウの幼虫はアブラナ科の植物であるキャベツが生合成するシニグリン(sinigrin)を食べると,その代謝産物が摂食刺激となりさらにキャベツを食べるようになる.一方,シニグリンはヒトに対し消化促進作用や利尿作用などのあることが報告されている.種々の機器による分析からこのような代謝産物の同定が行われており,質量分析法を中心とした分析技術により組織や細胞におけるすべての代謝産物を同定しようという試みが進展している.取得された質量スペクトルのピークを化学構造と関係づけて体系的に整理した"質量スペクトル-化学構造関係データベース"の構築により,新規の試料から得られた質量スペクトルから代謝産物を推定することが容易になれば,代謝産物を起点としたオミックス生物学は進展するであろう.そこで,このプロジェクトでは,メタボローム情報として取得される質量スペクトルから,同定あるいは未同定にかかわらず生物種や組織ごとにデータベースを構築し,メタボローム情報,とりわけ,生物種や組織,細胞に特異的に蓄積される二次代謝産物の情報について,その下流にある生物活性の情報と上流にあるゲノム情報とをリンクさせるインフォマティクスの基盤を構築することを目標とする.これにより,オミックス研究の上流から最下流まで生命活動を総合的に理解するために必要なデータベースがはじめて出揃うことになり,ポストゲノム科学の進展にあたえるインパクトは大きく,また,社会あるいは研究者へのニーズにも応えられるものと考えている.
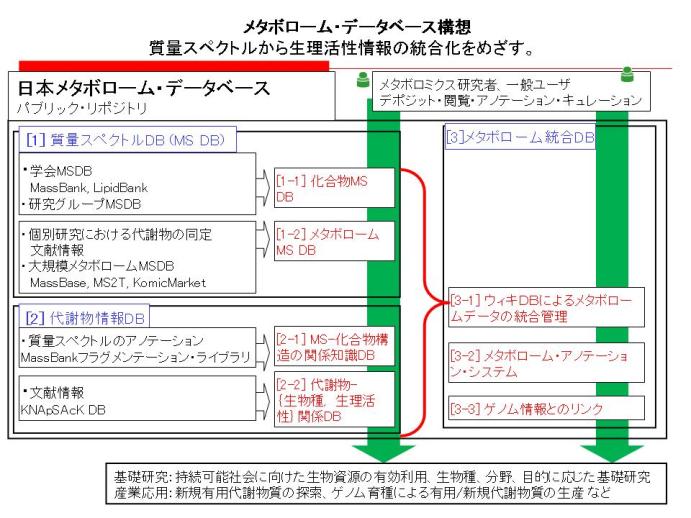
具体的には,質量分析により得られたメタボロームの質量スペクトルの情報を,化学構造の情報,また,生物種・時期・組織特異性などの生物情報と関連づけることをめざした日本メタボロームデータベースMetabolomics.JPを研究開発し構築する(図2).すなわち,① MassBankプロジェクトで開発された化合物および質量スペクトルのデータベースの技術基盤を活用し,生物試料を対象にした質量スペクトルデータベースを構築する.② MassBankプロジェクトで構築された質量スペクトル-化学構造関係データベースと生物種-代謝産物関係データベースのデータの拡充を図り,代謝産物と生理活性の関係データベースを新規に開発する.これらのデータベースをもとに代謝産物情報データベースを構築する.③ メタボロミクス研究者によるメタボロミクスの知識の集約,ならびに,生物試料の質量スペクトルに対する代謝産物情報のアノテーションの効率化を目的に,WebブラウザからWebページの発行や編集などが行えるWiki技術によりすべてのコンテンツを統合管理する技術を研究開発し実装する.
以下,これまで構築が進められているデータベースの特徴を概説する(表1).
1.MassBank
我が国は世界にさきがけて化学構造が既知のメタボロームの質量スペクトルについてのデータベースであるMassBankを構築しリーダーシップを執るにいたっている.それまで,物質の同定に不可欠な質量スペクトルについてのデータベースの構築が議論されていたにもかかわらず,質量分析の多様性への対応と,質量スペクトルの標準測定条件の確立,という2つの問題が解決されず,小規模なデータベースの分散的な構築にとどまっていた.MassBankはエレクトロスプレーイオン化-タンデム質量分析(ESI-MS2)による質量スペクトルのデータを化合物ごとに集約することにより,装置や測定条件に依存しない代謝産物の同定のための参照ESI-MS2データを開発することで,さきの2つの問題を解決した.現在では,メタボローム研究に必須のデータベースとしてその有用性が知られ国際的に広く利用されている.2011年11月現在,19研究グループ(日本15,米国2,ドイツ1,中国1)が11,614化合物について測定した30,312件のデータを8つのサーバから公開している.さらに,MassBankは生命科学データベースとしてはめずらしいデータ分散型データベースの実用化に成功している[1]. データベースの維持管理をプロジェクトに参加する研究者が負担することにより,低コストな研究者コミュニティデータベースを実現した.
2.MassBase/KomicMarket
かずさDNA研究所では,これまでに新産業の創出を目標に実用植物を中心とした微生物,動物を含む数百種の生物から得た生物試料について質量分析を行い,アーカイブとして独自のデータベースMassBase/KomicMarketより公開している[2].また,各種の質量分析装置を用いた生体からのメタボロームデータの大量取得ならびにメタボロームインフォマティクス技術の開発を進めており,総計で6万件以上の質量分析をすでに完了し,さらなるデータベースの開発を進めている.
3.KNApSAcK
メタボロームMSプロジェクトでは,質量スペクトルにより得られる精密分子量から既知の代謝産物の情報を得る目的で,科学文献をもとに生物種-代謝産物の情報を抽出しデータベースKNApSAcKを構築した[3].このデータベースは5万種の代謝産物につき10万対の生物種-代謝産物の関係を整理して公開しており,シロイヌナズナ国際コンソーシアム(The Arabidopsis Information Resource)の標準データベースとして認知されている.
4.Metabolomics.JP
これまで述べたデータベース,ならびに,そこで培われたノウハウを集約して公開できれば,我が国はまさにメタボロミクス研究における統合データベースの世界拠点をもつことになる.さらに,Wiki技術[4][5] をもとに,すでにスタートした日本メタボロームデータベースMetabolomics.JPを分子構造やクロマトグラム,生物活性の情報にまで広げ,真に学際的なデータベースとして統合する.国内の複数の学会による公式データベースを統合し公開することで,我が国の科学技術力とその方向性を世界にアピールできると信じて"メタボロームデータベース"の開発を進めている.
おわりに:オミックス情報の統合をめざして
ゲノムは生物の設計図である.しかし,生物が生きている状態は,時々刻々と動的に変化するトランスクリプトーム,プロテオーム,そして,最終的にはメタボロームの情報により把握される.地球上の植物により生合成される二次代謝産物の総数はおおまかに100万種と推定されており,このような膨大な数をつうじて生物間の相互作用がなされている.代謝産物の測定は質量分析などの機器分析にたよらなければならず,そこで得られたスペクトルの情報から代謝産物を的確に推定することができれば,たとえば,ヒトの健康維持のためのメタボロミクスのみならず,二次代謝産物をつうじた生物間の相互作用にもとづいた生態学への展開,さらには,これらの知見をもとにした持続可能な社会の構築へ適用も可能となるだろう.このようなメタボロミクスを入り口としたオミックス研究(図1)を目標として,このプロジェクトではメタボロミクスにおける知の集積および統合を図りたいと考えている.
参考文献
- Horai, H., Arita, M., Kanaya, S. et al.: MassBank: a public repository for sharing mass spectral data for life sciences. J. Mass Spectrom., 45, 703-714 (2010) ↑
- Iijima, Y., Nakamura, Y., Ogata, Y. et al.: Metabolite annotations based on the integration of mass spectral information. Plant J., 54, 949-962 (2008) ↑
- Shinbo, Y., Nakamura, Y., Altaf-Ul-Amin, M. et al.: KNApSAcK: A comprehensive species-metabolite relationship database. in Plant Metabolomics (Saito, K., Dixon, R. A., Willmitzer, L. eds), pp. 165-184, Springer Verlag, Heidelberg (2006) ↑
- 時松敏明, 有田正規: 代謝マップビューワで見るフラボノイド. 細胞工学, 25, 1388-1393 (2006) ↑
- Arita, M.: A pitfall of wiki solution for biological databases. Brief. Bioinform., 10, 295-296 (2009) ↑
↑ 押下で本文に戻ります。
表説明
| データベース名 | URL | 概説 |
|---|---|---|
| KNApSAcK Family | http://kanaya.naist.jp/KNApSAcK_Family/ | 生物種ー代謝産物関係データベース,世界の薬用植物データベース,漢方生薬データベース,インドネシア薬用植物データベース,生物ー生物活性関係データベースのメインページ |
| KomicMarket | http://webs2.kazusa.or.jp/komics/ | ピークのアノテーションあるいはキュレーションが行われたメタボローム情報を公開するためのデータベース |
| MassBank | http://www.massbank.jp/ | 代謝産物に関連する化合物について測定された質量スペクトルの公的なレポジトリ.日本質量分析学会の公式データベース |
| MassBase | http://webs2.kazusa.or.jp/massbase/ | シロイヌナズナと実用植物,動物などのメタボロームプロファイルデータのアーカイブ |
| Metabolomics.JP | http://metabolomics.jp/ | フラボノイドなどさまざまな二時代謝産物の情報を収録 |

Licensed under a Creative Commons 表示2.1日本 license ©2012 金谷重彦(奈良先端科学技術大学院大学)、西岡孝明(奈良先端科学技術大学院大学)、有田正規(理化学研究所)、櫻井望(かずさDNA研究所)
なお、本記事は細胞工学2012年1月号掲載の原稿を改変したものです。