第4回「微生物統合データベース"MicrobeDB.JP"」
はじめに
数学的な基盤の乏しいライフサイエンス分野において,知識の集積体であるデータベースは知識の参照のみにとどまるものではなく,新たな研究分野を切り拓くうえで欠くことのできないきわめて重要な研究基盤である.
ライフサイエンス分野のなかでも微生物の研究の歴史は古く,これまでに蓄積された知識は分類学的な情報,菌株保存情報にはじまり,各種のオミックス情報,メタゲノム情報など膨大かつ多岐にわたっている.微生物の研究においては研究者ごとの分野の特異性が高く,自らの研究対象の外に興味の範囲を拡大する機会はまれであったため,ゲノム情報の蓄積が進んだ現在でも通常の微生物研究者がこれら膨大な情報を横断的に活用する動きはあまり一般的ではなかった.しかし,近年,新型シークエンサーに代表される実験機器類の飛躍的なハイスループット化により,ゲノムサイズの小さい微生物は比較的容易にゲノム解析が可能であることから,これまでゲノム情報には疎遠であった微生物研究者が自らゲノム解析を行うケースも増えてきており状況は変わりつつある.また,これまで1種類のゲノム解析にとどまっていた研究も,いちどに複数種のゲノムを解読し比較ゲノム解析により新たな知見を得るという"データ駆動型"の大規模な研究へと発展しつつある.さらに,環境中の微生物集団を丸ごとゲノム解析するメタゲノム解析も実現し,海洋や土壌など自然環境だけでなく,ヒトの口腔内,腸内,皮膚に存在する微生物集団を対象とした解析も急速な勢いで進んでいる.したがって,これらゲノムやメタゲノムなど圧倒的な量のデータを横断的にかつ簡便に利用できれば,新たな仮説や研究分野の創出がより容易になるものと期待される.
しかしながら,この爆発的に生み出される情報は多様かつ膨大であり,また,細分化されているため,微生物学に革新的な進展をもたらす可能性をもっているにもかかわらず,個々の微生物研究者やコミュニティが自らの研究のため活用することはますます困難になってきている.これはまさにデータベースのシステムにおける問題であり,効果的な統合データベースの構築によりこの状況を改善し大きな進展をもたらすことが期待できる.さまざまなデータが横断的に統合され容易に利用可能な環境となれば,たとえば,これまで病原菌にしか興味のなかった研究者も分野横断的な検索により研究対象の微生物がほかの環境ではどのような挙動を示すのかといった幅広い情報を得ることができ,その結果,従来の常識にとらわれない画期的な発見をもたらす可能性もある.
1.微生物統合データベースの開発
そこで筆者らは,ゲノム情報を核としてさまざまな微生物学における知識を統合し,幅広い分野での微生物学の発展に資することのできる"微生物エンサイクロペディア"の構築を目標とした研究課題に取り組でいる.この研究開発では,我が国をはじめ世界中に散在している細菌(真正細菌,古細菌,バクテリオファージ)におけるゲノムやトランスクリプトームなどの各種のオミックス情報やメタゲノム情報を徹底的に収集し,遺伝子機能,分類学的な情報,菌株保存の情報,表現型の情報(病原性,環境)など膨大な知識を適切に整理したうえでゲノム情報を核として統合する.
国内外に散在している個々のデータベースはそれぞれが異なるプロジェクトにより独立に構築また更新されているため,このことがデータ分散化の原因となってデータベースの統合化および横断的な利用を困難としてきた(図1 a,b).たとえば,菌株保存に関するデータベースはそれぞれの菌株保存センターや研究グループにより国内外に複数が存在しそれぞれのグループで維持および更新がなされている(図2).これらの菌株の情報ならびに培養条件の情報などをゲノムやメタゲノムなどさまざまなオミックスデータとともに統合できれば,データベース横断的に検索するだけで新たな研究シーズを生み出すことも可能になると期待できる.
分散化している多様なデータベースを1箇所に集約し維持および更新していくのは非現実的であるが,データベースを分散化したまま横断的に利用を可能とするのが急速に発展しつつあるセマンティックウェブ技術である(図1c).その技術面での詳細は,本シリーズ第2回「データベースを統合利用するための基盤としてのセマンティックウェブ技術」(URL:https://events.biosciencedbc.jp/article/02)を参照されたい.微生物統合データベースではこのセマンティックウェブ技術を積極的に取り入れることにより統合化をはかり,分散したデータベースを横断的に利用可能とすることを目標としている.また,データベースの構築のみならず,データベースを利用して新たな知見を得るための解析システムの開発も同時に進めている.この解析システムは大規模なデータ生産者に対してこのデータベースシステムを積極的に利用する価値を提供し,大規模データを集約する駆動力となることが期待できる.大規模データが集約できればデータ間の連携が加速度的に進み,より実用性の高いデータベースシステムに発展することも期待できる.
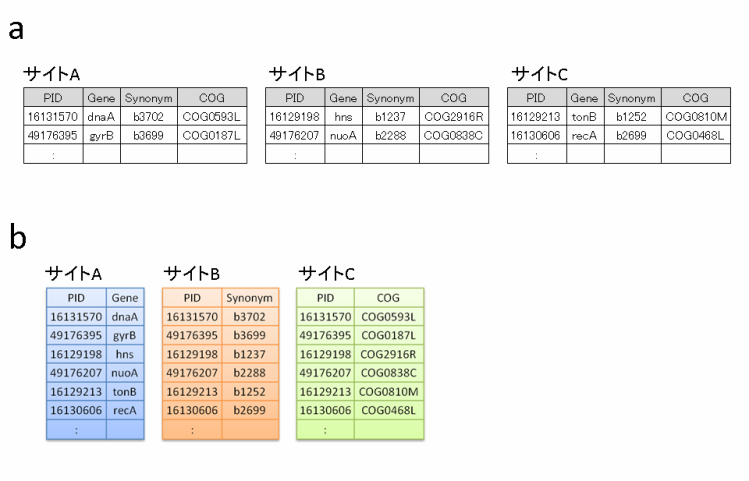
図1.分散化されたデータベースとセマンティックウェブ技術
(a)レコードにより分散化されたデータベースの概念図.サイトA,サイトB,サイトCのそれぞれ同一の形式によりデータが蓄積されている.これらを統合する場合,各サイトにおいて簡単にフィールドを追加したり変更したりすることはできない.
(b)フィールドにより分散化されたデータベースの概念図.それぞれのサイトで蓄積しているデータの種類が異なっている.この場合,新たなレコードの追加は困難である.
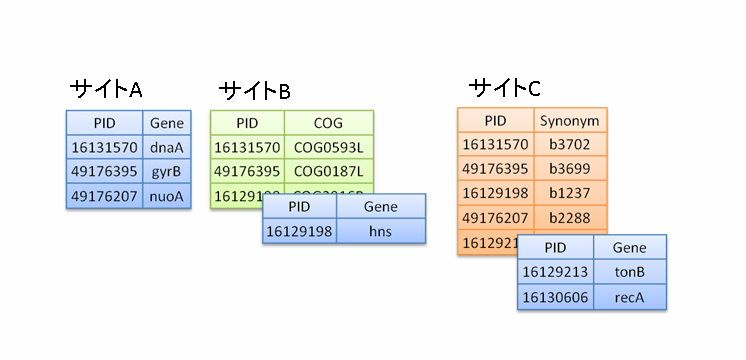
図1.分散化されたデータベースとセマンティックウェブ技術
(c)レコード,フィールドともに分散化されたデータベースの概念図.セマンティックウェブではすべてのサイトにおいてレコードの情報,フィールドの情報,その値という3つ組によりデータを保存することで,それぞれのサイトにおいて独自に更新や追加などを可能としつつ統合化することが可能になる.
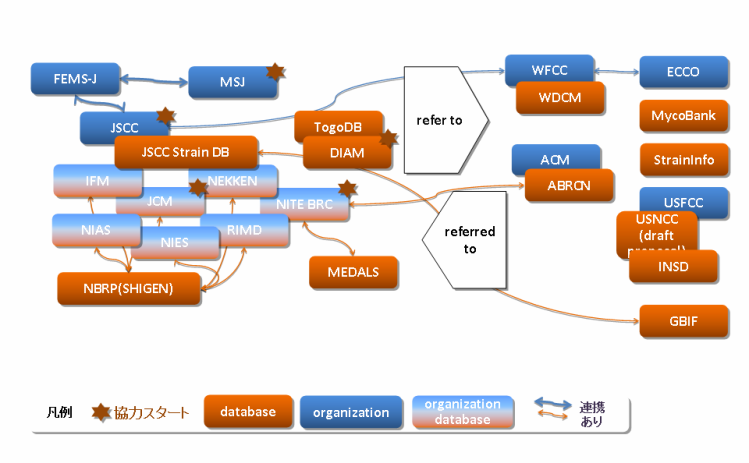
図2.菌株保存データベースのネットワーク
*印は微生物統合データベースプロジェクトにて統合化を開始したもの.
- ABRCN:Asian Biological Resource Center Network
- ACM:Asian Consortium for the Conservation and Sustainable Use of Microbial Resources
- DIAM:Data Biosafety for the Industrial Application of Microbes
- ECCO:European Culture Collections' Organization
- FEMS-J:Federation of Microbiological Society of Japan
- GBIF:Global Biodiversity Information Facility
- IFM:千葉大学真菌医学研究センター
- INSD:DDBJ/EMBL/GenBank
- JCM:Japan Collection of Microorganisms
- JSCC:Japan Society for Culture Collections
- MSJ:the Mycological Society for Japan
- MycoBank:MycoBank, the fungal website
- NBRP:National BioResource Project
- NEKKEN:長崎大学熱帯医学研究所
- NIAS:農業生物資源研究所ジーンバンク微生物遺伝資源部門
- NIES:国立環境研究所微生物系統保存施設
- NITE BRC:製品評価技術基盤機構 Biological Resource Center
- RIMD:大阪大学微生物病研究所感染症国際研究センター病原微生物資源室
- StrainInfo:http://www.straininfo.net/
- USCCN:US Culture Collection Network
- USFCC:US Federation for Culture Collections
- WDCM:WFCC-MIRCEN World Data Center for Micoorganisms
- WFCC:World Federation for Culture Collections
2.微生物統合データベースの展望
微生物統合データベースでは,これまで述べたとおり,ゲノム情報を核としてさまざまな微生物学における知識を統合し,幅広い分野での微生物学の発展に資することのできる"微生物エンサイクロペディア"の構築をめざしている.微生物データの統合化は微生物の体系的な理解を促進し,これまでの"仮説検証型"の研究のみならず,膨大なデータのなかから新たな仮説を導く"データ駆動型"の研究という新たな方向性を強力に推進することを可能にする.
その際に重要となるのが,さきに述べたセマンティックウェブ技術である.セマンティックウェブ技術がめざすところはW3CによるOWL Web Ontology Language Guide(URL:http://www.w3.org/TR/owl-guide)から類推することができる.このガイドの冒頭にはデータベース検索における問い合わせ文として "Tell me what wines I should buy to serve with each course of the following menu. And, by the way, I don't like Sauternes." という例があげられており,この問い合わせ文による検索に対し適切な結果を出力することがセマンティックウェブ技術の目標のひとつとなっている.インターネットにおける初期のWeb検索エンジンでは,たとえば"東京駅周辺のおいしいラーメン屋"を検索したい場合,検索者がラーメン屋の屋号を知識としてもっていないとスムースな検索が困難であった.ところが,現在よく利用されている検索エンジンでは"東京駅,おいしい,ラーメン"という検索語でよい検索結果を得ることが可能になっており,おいしいラーメン屋の屋号を知らない人でもめざすラーメン屋の情報を簡単に得ることができるようになっている.それでは,ライフサイエンス分野ではどうだろうか.多くのデータベースにおける検索では,さきのような初期の検索エンジンと同様,たとえば"gyrB"といった専門用語を知らないかぎり検索そのものが困難になっている.これは,現在のライフサイエンス分野におけるデータベースのほとんどが遺伝子名など"もの"(データ)を蓄積したデータベースであって,たとえば遺伝子間相互作用など"こと"(意味)に関する記述が実現できていないことに起因する.複数の"もの"の情報から"こと"に関する情報を得るにはきわめて高い知識レベルにもとづく高度な知的作業が要求されるため,専門家以外がそのデータベースから有益な情報を得ることは困難である.セマンティックウェブ技術により多様なデータベースを統合化できれば,近い将来,"大腸菌をLB培地により37℃で培養した際に発現量の高い遺伝子"というような問い合わせ文に対して高発現量の遺伝子の一覧を出力することも可能になるだろう.さらに,これら情報を利用者が意識することなく収集できれば,利用者の興味の範囲をこえた情報も同時に得ることができ,さらなる研究の発展が期待できる.
微生物は,海洋や河川,土壌や大気,さらには,ヒトや動物の腸内,皮膚,口腔内など地球上のあらゆる環境に存在し,その環境に特化した多様な微生物が群集 を形成し棲息することで地球環境における物質循環の根幹を形成しているといっても過言ではない.したがって,環境の根幹を形成する微生物群集のそれぞれの個体および総体としての生命システムを明らかにするためには,そこにある遺伝子など"もの"を理解するのみならず,それらがどのようなしくみで連携しているかなど"こと"に関する理解が必須となる.したがって,セマンティックウェブ技術にもとづくデータベースの統合化は今後の微生物学分野の発展に欠くことのできない課題となっている.さらに,微生物とさまざまな高等生物との相互作用に着目すれば,近い将来,より高等な生物のデータベースとの統合化も必須となるだろう.たとえば,植物データベースと光合成細菌,菌根菌,エンドファイト,植物病原菌などのデータベースとの統合化は次世代農業の基盤となりうるだろうし,ヒトの免疫と細菌との関係性の観点から,ヒトメタゲノム研究における究極のメタデータとしてヒトゲノムのデータを微生物データベースに統合することもパーソナルゲノムとニュートリゲノミクスの融合に大いに貢献するだろう.
おわりに
新型シークエンサーに代表される実験機器の飛躍的なハイスループット化は,今後,ますます加速され,ゲノム研究やメタゲノム研究など"データ駆動型"の研究がいよいよ本格化すると考えられる.さらに,データ生産技術の発展を考えれば,プロテオームやメタボロームのデータについても将来の技術革新により配列情報と同様に情報爆発を起こす可能性があり,これらのデータも微生物統合データベースを核として可能なかぎり統合していきたいと考えている.これまでブラックボックス化していた微生物の生命システムを統合化データベースを利用した"データ駆動型"研究により明らかにすることは,工業,建築,農業,畜産業や医薬,健康,宇宙など,微生物が関与する広範な研究分野あるいは産業にとり最重要な研究課題のひとつとなりえるだろう.
- 東工大グループ:森 宙史,吉野弘二,竹原潤一,小西史一,黒川 顕
- 遺伝研・DDBJグループ:藤澤貴智,神沼英里,菅原秀明,中村保一
- 基生研グループ:千葉啓和,西出浩世,内山郁夫
- DBCLSグループ:岡本 忍,川島秀一,川本祥子,片山秀明,山本泰智

Licensed under a Creative Commons 表示2.1日本 license ©2012 黒川顕(東京工業大学大学院)
なお、本記事は細胞工学2011年12月号掲載の原稿を改変したものです。