第2回「データベースを統合利用するための基盤としてのセマンティックウェブ技術」
はじめに
ライフサイエンス分野の研究により生み出される多様かつ膨大なデータから必要な情報を効率的に得るためには,ばらばらに構築されているデータベースを統合的に扱うための情報基盤の構築が必要不可欠である.連載第1回「データベースの現状と未来」(https://events.biosciencedbc.jp/article/01)では,データベース統合化のための具体的なステップとして,つぎの3つの段階があげられた.
- 第1段階:データベースを網羅的に収集しメタデータを付与すること
- 第2段階:それぞれのデータベースにおいてフォーマットと用語の統一を行うこと
- 第3段階:複数のデータベースを再構築し使いやすいインターフェイスにまとめあげること
大学共同利用機関法人 情報・システム研究機構 ライフサイエンス統合データベースセンター(DBCLS:Database Center for Life Science,URL:http://dbcls.rois.ac.jp/)では,このうちの第3段階をスムーズに実現することを目標として,現在,セマンティックウェブを利用した第2段階の技術開発を進めている.ここでは,その基幹技術であるRDF(resource description framework)について紹介する.
1.RDFとは
セマンティックウェブとは英国の計算機科学者Tim Berners-Leeにより提唱された概念で,ウェブページに書かれている情報の意味をコンピューターにも扱えるよう記述することにより自動的な情報の収集と利用を容易にしようとするものである.もしこれがうまくいけば,人工知能があなたの質問にウェブに存在するすべてのデータから的確に答えてくれる時代が来るかもしれない.
そんなセマンティックウェブの基幹技術のひとつとして,情報記述の規格RDFがあげられる.RDFでは,すべての情報を主語・述語・目的語からなる三つ組という単純なデータ構造により表現する.主語は必ずURI(uniform resource identifier,ウェブ上のアドレスであるURLなどを包括する概念)であり,たとえば,あるタンパク質を記載したウェブページのURLなどとなる.目的語は主語を説明するデータ(文字列や数値など),あるいは,ほかへのリンクを表すURIである.述語は主語と目的語との関係(たとえば,結合する,配列をもつ,など)を表し,統一された用語が使われる.
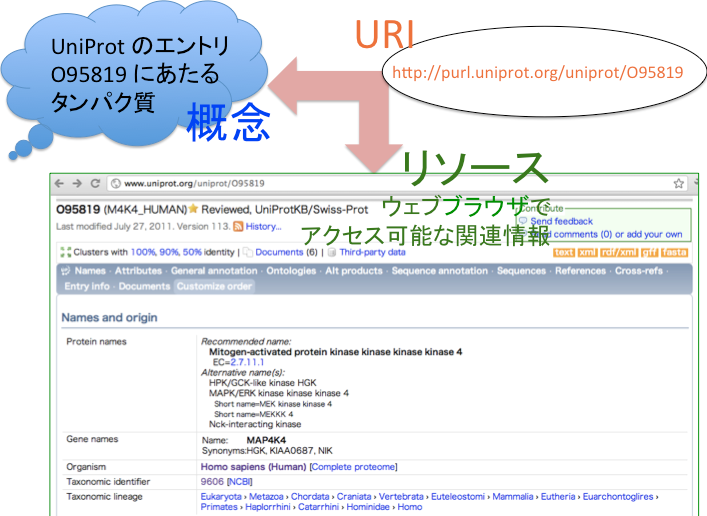
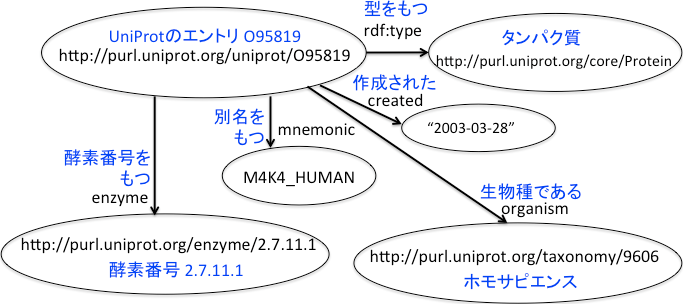
広く使われているタンパク質のデータベースUniProt(URL:http://www.uniprot.org/)では,すでに数年前からすべてのデータをRDFにより管理するようになっている.たとえば,図1のように,"UniProtのエントリO95819にあたるタンパク質"は"http://www.uniprot.org/uniprot/O95819"というURIとしてコンピューターにおいて一意に識別される.UniProtの場合,人間もこれをURLとしてウェブブラウザーで開けば関連する情報がみられるようになっている. 図2は,UniProtのエントリO95819に関する情報の一部をRDFで表現した概要図である.ここから,RDFで表現されたデータは表形式ではなく,主語・述語・目的語の三つ組からなるネットワーク(グラフ形式)になっていることがわかる.
このシンプルな三つ組によるグラフ形式は柔軟なデータベースの構築にはとても有効で,実際,UniProtでは扱うデータの多様性に追従することが既存のデータベースシステムでは困難になってきたためRDFを採用したといわれている.
図2.UniProtのエントリO95819についてRDFで表現した周辺情報
ここに表現された情報は,それぞれ,
- "http://www.uniprot.org/uniprot/O95819はタンパク質である"
- "http://www.uniprot.org/uniprot/O95819は2003年3月28日に作成された"
- "http://www.uniprot.org/uniprot/O95819の生物種はホモサピエンスである"
- "http://www.uniprot.org/uniprot/O95819はM4K4_HUMANという別名をもつ"
- "http://www.uniprot.org/uniprot/O95819は酵素番号2.7.11.1をもつ"
ことを意味する.
2.データベース統合化のステップ
ライフサイエンス統合データベースセンターがRDF関連技術を用いて,データベース統合化の3つの段階に対しどのような取り組みを行っているかを紹介する[1].
第1段階:データベースを網羅的に収集しメタデータを付与すること
現在,バイオサイエンスデータベースセンター(NBDC:National Bioscience Database Center,URL:http://biosciencedbc.jp/)において公開されている生命科学系データベースカタログ(URL:http://biosciencedbc.jp/dbcatalog/) は,世界に散在するデータベースの情報を集積しており,対象や生物種などの項目別に閲覧することができる.このとき,データベースの概要・URL・提供機 関・対象生物種・データ種別・稼働状況などがメタデータとして付与されていることで利用者の目的に応じた検索ができるようになっている.海外に目をむける と,ゲノム情報のアノテーションを行う専門家による国際団体International Society for Biocuration(ISB,URL:http://biocurator.org/)では,データベースに統一的なメタデータやライセンスの表記がないと遺伝子のアノテーションなどに利用しにくいことから,BioDBCore(URL:http://biocurator.org/biodbcore.shtml)とよばれるガイドラインの制定を進めている.
RDFはウェブページに書かれている情報の意味をコンピューターで扱うための技術として出発した.これをデータベースに置き換えて考えると,データベース に書かれている情報の意味,すなわち,メタデータを扱うにはやはりRDFがむいている.このため,ライフサイエンス統合データベースセンターでも BioDBCoreと協力しつつメタデータの標準化とRDF化を進めている.
第2段階:それぞれのデータベースにおいてフォーマットと用語の統一を行うこと
さまざまなデータベースを統合的に扱うにあたり,これまでは,新しくデータベースが増えた場合,まず内容をみて何が記述されているかを調べ,そのフォー マットに応じて統合データベースの設計を見直す必要があった.また,追加したデータベースを統合的に利用するためには,既存のデータベースとの対応を調査 し,対応表をつくって関連づけを行う必要があった.このような個別の対応と既存のシステムとの統合は,可能ではあるものの,頻繁にソフトウェアを書き直す 必要があるなど多大な労力がかかる.
RDFを利用した場合は新しい種類のデータを追加するにあたり既存のデータベースに手をくわえる必要はなく,新しいデータを主語・述語・目的語の三つ組と して統合データベースに足すだけでよい.もちろん,この変換にはこれまでどおり,もとのデータのフォーマットの解釈は必要であるが,とにかくRDFになっ てしまえば既存のデータベースの設計見直しは不要となる.さらに,すべてのデータがRDFとして統一されるため,個々のデータの由来やフォーマットによる 制限がなくなり,関連するすべてのデータをシームレスに取得することができるようになる.
〈URIとオントロジー〉
ここでキーとなるのがURIである.異なるデータベースに由来するデータであっても,同じ概念に同じURIが利用されていればRDFのデータそのものが自動的に対応づけられる.具体的には,あるタンパク質MEKKK4をさすURIがアミノ酸配列データベースと立体構造データベースとで共有されていれば,データベースを統合したときこの2つは自動的に関連づけられる,というイメージである.実際には,RDFを作成する際のURIの割り当ての方法について制限がないため,同じ概念に同じURIが割り当てられることは必ずしも期待できない.しかし,2つのデータベースで使われているURIのあいだの対応関係を,やはりRDFとして追加しておけばそれですむ.これは,データベースの統合におけるRDFの利用の大きなメリットといえ,データベースの更新のたびにソフトウェアを書き直す必要性が大きく減少する(図3).
さらに,RDFを利用したセマンティックウェブの世界では,より高度な概念での統合利用も期待されている.単純にURIどうしが一対一に対応する場合だけでなく,概念的に関連はあるがまったく同一のものではない場合にも,それらの関係を表す述語を利用することで関連する情報をひきだすことができるのである.さきほどのタンパク質MEKKK4の例でいえば,類似するほかのキナーゼを列挙するとか,MEKKK4によりリン酸化される基質を探すとかいった検索をイメージするとよいかもしれない.
このために必要となってくるのが,関係性を表す述語の意味を明確にして統一することである.このような用語の整理と意味づけのなされたものはオントロジーとよばれており,Gene Ontology(GO)などが広く利用されている.オントロジーの整備については,Open Biological and Biomedical Ontologies(OBO,URL:http://www.obofoundry.org/)などの組織で精力的に進められており,オントロジーを集積するレポジトリーとして,米国National Center for Biomedical OntologyによりBioPortal(URL:http://bioportal.bioontology.org/)が構築されている.
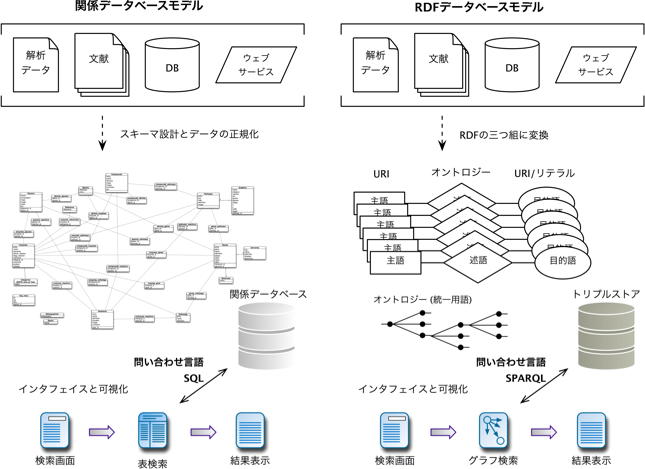
図3.RDFを利用したデータベースの統合
従来の関係データベースによるデータベースの統合(左)と,RDFデータベースモデルによるデータベースの統合(右)を比較して示した.関係データベースモデルにおいて必要なスキーマ(データベース構造において,表と表との関係や表のなかのどの列にどのようなデータを格納するかを定義するもの)の設計をデータの正規化が,RDFデータベースモデルでは不要となる.
〈Linked Data〉
近年,さまざまなデータベースのプロバイダーがRDFによるデータの記述を試みている.実のところ,論理的にきっちりとしたオントロジーを整備するにはその分野の専門家による調査と議論が必要で,その完成までに時間もかかる.また,いちどつくられたオントロジーも新しい知見をもとに改訂する必要のあることが多く,それがおわるのを待っていてはいつまでもRDFをつくりはじめることができないという側面もある.一方で,RDFには異種多様なデータをつないでいくだけでも実用的には十分なメリットがあるため,まずは,つながるデータをできるかぎりつなげてRDFとして公開してしまおうというムーブメントが起こってきた.この試みはLinked Dataとよばれており,さまざまなデータが相互に連結したひとつの巨大なグラフがつくられるようになってきている.もちろん,これらLinked Dataにはライフサイエンス分野のデータも含まれている.UniProtが自らのデータベースをRDF化して公開したことを皮切りに,Bio2RDF(URL:http://bio2rdf.org/)がサードパーティとして四十数個の主要なライフサイエンスのデータベースをRDF化したことにより,ライフサイエンス分野のデータはLinked Dataのなかでも大きな位置をしめるようになった. 図4は,公開されているRDFデータを収集するプロジェクトLinking Open Data(URL:http://www.w3.org/wiki/SweoIG/TaskForces/CommunityProjects/LinkingOpenData)により集計されたデータベースのあいだのリンクの状況を示したものである.Wikipedia内の構造化されたデータから作成されたDBPedia(URL:http://dbpedia.org/)を中心として,さまざまな種類のデータが互いに結ばれていることがわかる.このうち,ピンク色で示した部分がライフサイエンス関係のデータベースであり,巨大なデータ空間のなかライフサイエンス分野のデータが大きく存在していることを読み取ることができる.
図4.Linking Open Dataで集められたRDF化されたデータベースにおけるリンクの状況
ライフサイエンス関係のデータベースをピンク色で示す.2011年9月現在.
Linking Open Data cloud diagram, CC-BY-SA:Richard Cyganiak and Anja Jentzsch, http://lod-cloud.net/
〈ライフサイエンス統合データベースセンターにおける技術開発〉
ライフサイエンス統合データベースセンターでは,RDFとして利用できるデータを充実させるため国内のデータベースのプロバイダーに対しRDF化の支援を行っている.とくに,DDBJ(日本DNAデータバンク,URL:http://www.ddbj.nig.ac.jp/)とPDBj(日本蛋白質構造データバンク,URL:http://www.pdbj.org/index_j.html) については,構築者と協力してRDF化を進めている.また,これまで統合化のために受け入れてきたデータベースや,統合の観点から有用性あるいは再利用性 の高いと思われるデータベースに対しては,ライフサイエンス統合データベースセンターにおいてRDF化を行う予定であり,また,その経験をもとにRDF化 のガイドラインを作成しRDF化技術の普及をめざしたいと考えている.
ところで,作成されたRDFはただの三つ組の羅列なので,そのままでは利用しにくい.そこで利用されるのが,三つ組(トリプル)を格納(ストア)する RDFデータ専用のデータベース,トリプルストアである.代表的なトリプルストアとして,Virtuoso,4store,OWLIM,Mulgaraな どがあるが,これらの性能はデータの量や問い合わせの複雑さにより大きく異なることが知られ,パソコンで扱える程度の少量のデータに対し高速な検索を実現 するものから,超大規模なデータを組み合わせた高度な検索を効率的に実現するシステムまで,それぞれに特色がある.ライフサイエンス統合データベースセン ターでは,ライフサイエンス分野のデータの特徴とトリプルストアの長短との関係を明確にするため,さまざまなトリプルストアの性能の比較を行っている.こ の調査結果をもとに,統合データベースに適したトリプルストアを選択もしくは開発する予定である.
第3段階:複数のデータベースを再構築し使いやすいインターフェイスにまとめあげること
さて,RDFとしてフォーマットが統一され,すべてをトリプルストアに格納した統合データベースができたところで,ユーザーはこれをどのように利用するこ とができるのだろうか.トリプルストアの検索にはSPARQLという標準仕様言語が使われるが,一般の利用者にとりSPARQLを使って問い合わせを記述 するのはむずかしい.そのため,トリプルストアに集積されたデータをもとに,目的に応じた使いやすいインターフェイスや可視化の方法を開発する必要がある.
ライフサイエンス統合データベースセンターでは,蓄積された多種多様なデータをユーザーにどのように提示するか,また,そのデータをやりとりする適切なイ ンターフェイスは何かを,ユースケースをつうじ検討している.とくに,データの検索と解析ワークフローの構築に重点をおき,データの検索については,検索 されたデータのうちオントロジーなどを活用してどの項目をどのように表示するべきか,解析ワークフローの構築については,ツールのあいだでどのようにデー タをやりとりすべきかを議論している.今後は,これらの検討にもとづいて統合データベースのプロトタイプの作成を行い,その有効性を確かめていく予定であ る.
おわりに
ここでは,RDFを用いたデータベース統合と,それにむけたライフサイエンス統合データベースセンターの取り組みについて述べた.RDFを用いた統合デー タベースを有用なものとするためには,まずはできるだけ多くのデータをRDFとして扱えるようにする必要がある.今後は,さまざまなデータベースのRDF 化を推進するとともに,ユーザーがRDF化のメリットを享受できるようなシステムの開発を進めていきたい.
参考文献
- 片山俊明: バイオインフォマティクスのソフトウェアとサービスにおける標準化. 実験医学, 29, 174-181 (2011) ↑
↑ 押下で本文に戻ります。

Licensed under a Creative Commons 表示2.1日本 license ©2011 山口敦子(ライフサイエンス統合データベースセンター)、片山俊明(東京大学医科学研究所)
なお、本記事は細胞工学2011年11月号掲載の原稿を改変したものです。